Journal de Ciencias de la Salud
Palabras clave
Almacenamiento digital de datos; Ácido desoxirribonucleico (ADN); Binario; Codificación; Descodificación; Secuenciación
Introducción
Hoy en día, es una era explosiva de los grandes datos. Estos big data existen y cubren casi todos los lugares, desde las tiendas de comestibles hasta los bancos, desde fuera de línea hasta en línea, desde la academia hasta la industria, desde el hospital hasta la comunidad, desde la organización hasta el gobierno. El almacenamiento y la gestión de los big data se están convirtiendo en una seria preocupación. En la actualidad, la mayor parte de los datos de todo el mundo se almacenan principalmente en soportes magnéticos y ópticos, como discos duros, CDs, cintas, DVDs, discos duros portátiles y memorias USB. Sin embargo, la velocidad de crecimiento de estos datos de archivo aumenta explosivamente a un ritmo exponencial. Estos medios tradicionales y su limitada capacidad de almacenamiento de datos no pueden satisfacer las necesidades del rápido aumento de los datos digitales. Además, la durabilidad del almacenamiento de datos de estos medios es un reto importante. Su durabilidad es muy limitada. Estos soportes sólo duran un tiempo muy limitado. Por ejemplo, los discos pueden durar varios años y las cintas varias décadas. Otros soportes electrónicos pueden conservarse en buen estado durante varias décadas. La capacidad de almacenamiento de datos es otro problema para guardar grandes datos digitales. Un CD puede almacenar varios cientos de megabytes (MB) de datos. Un gran disco duro puede almacenar un par de terabytes (TB) de datos. Sin embargo, su capacidad está muy lejos de la exigencia de los explosivos datos de información.
Según dijo Patrizio, en 2018 hay en total 33 Zettabytes (ZBs) de datos en todo el mundo (https://www.networkworld.com), lo que equivale a 22 billones de gigabytes (GBs). Por lo tanto, se necesita una tecnología de almacenamiento novedosa y un sistema innovador para satisfacer los requisitos de esta era moderna. El ácido desoxirribonucleico (ADN), debido a sus ventajas únicas, se espera que sea un medio ideal para el almacenamiento de datos digitales. Almacenar los datos digitales en el ADN no es una historia nueva. En realidad, fue descrito por el físico soviético Mikhail Neiman en la década de 1960 (https:// www.geneticsdigest.com). Sin embargo, fue la primera vez que se demostró que el ADN puede almacenar datos digitales en 1988. Aquí, en primer lugar, introducimos las aplicaciones del ADN como un nuevo medio de almacenamiento de datos digitales y a continuación discutiremos más detalles en este campo del ADN como medio de almacenamiento de datos.
Revisión de estudios anteriores
El sistema numérico binario
Los ordenadores y otros dispositivos electrónicos digitales almacenan datos y operan con el sistema numérico binario que utiliza sólo dos números digitales o 0 y 1 . Los textos se convierten en versión binaria en el sistema informático. A su vez, los ordenadores operan y calculan en binario, convirtiendo finalmente la información en textos legibles. Un byte contiene ocho bits formados por 0 ó 1 y tiene 28 (256) valores posibles (de 0 a 255), y almacena una sola letra (Figura 1 y Tabla 1) . Como se muestra en la tabla de conversión ASCII (Tabla 1). Las veintiséis letras con las mayúsculas y minúsculas se convierten entre Letra, Binario y Hexadecimal. Para almacenar un archivo o documento grande se necesita mucha más memoria de datos. Una canción normal puede necesitar docenas de megabytes, con un par de gigabytes para almacenar una película y varios terabytes para los libros almacenados en una gran biblioteca. Como se muestra en la Tabla 2 están los tamaños de medida y memoria para el uso del sistema binario desde la unidad más pequeña «byte» hasta las grandes unidades, incluyendo byte (B), kilobyte (KB), megabyte (MB), gigabyte (GB) terabyte (TB), pegabyte (PB), Exabyte (EB), zettabyte (ZB), yottabyte (YB), brontobyte (BB), Geopbyte (GPB), etc. (https://www.geeksforgeeks.org&https://whatsabyte.com). Las unidades como brontobyte (BB), Geopbyte (GPB) son valores enormes inimaginables que quizá nunca se utilicen en nuestro mundo real (Tabla 2).

Figura 1: La cadena de texto «Almacenamiento de datos digitales de ADN» se convirtió en bits binarios.
Table 1: The conversion ASCII table of the twenty-six letters with the upper and lower cases among letter, binary and hexadecimal.
| Letter | Binary | Hexadecimal | Letter | Binary | Hexadecimal |
|---|---|---|---|---|---|
| A | 1000001 | 41 | a | 1100001 | 61 |
| B | 1000010 | 42 | b | 1100010 | 62 |
| C | 1000011 | 43 | c | 1100011 | 63 |
| D | 1000100 | 44 | d | 1100100 | 64 |
| E | 1000101 | 45 | e | 1100101 | 65 |
| F | 1000110 | 46 | f | 1100110 | 66 |
| G | 1000111 | 47 | g | 1100111 | 67 |
| H | 1001000 | 48 | h | 1101000 | 68 |
| I | 1001001 | 49 | i | 1101001 | 69 |
| J | 1001010 | 4A | j | 1101010 | 6A |
| K | 1001011 | 4B | k | 1101011 | 6B |
| L | 1001100 | 4C | l | 1101100 | 6C |
| M | 1001101 | 4D | m | 1101101 | 6D |
| N | 1001110 | 4E | n | 1101110 | 6E |
| O | 1001111 | 4F | o | 1101111 | 6F |
| P | 1010000 | 50 | p | 1110000 | 70 |
| Q | 1010001 | 51 | q | 1110001 | 71 |
| R | 1010010 | 52 | r | 1110010 | 72 |
| S | 1010011 | 53 | s | 1110011 | 73 |
| T | 1010100 | 54 | t | 1110100 | 74 |
| U | 1010101 | 55 | u | 1110101 | 75 |
| V | 1010110 | 56 | v | 1110110 | 76 |
| W | 1010111 | 57 | w | 1110111 | 77 |
| X | 1011000 | 58 | x | 1111000 | 78 |
| Y | 1011001 | 59 | y | 1111001 | 79 |
| Z | 1011010 | 5A | z | 1111010 | 7A |
*Note: ACSII (American Standard Code for Information Interchange): serial digital codes to represent number, letters, numerals, and other symbols and to be used as a standard format in the computer system.
Table 2: The sizes of measurement and memory.
| Sizes | Byte Magnitude | Units | Storage* |
|---|---|---|---|
| 1 B | 100 | Byte | A character «A», «1», «$» |
| 10 B | 101 | ||
| 100 B | 102 | ||
| 1 KB | 103 | Kilo byte | The size for graphics of small websites ranges between 5 and 100 KB |
| 10 KB | 104 | ||
| 100 KB | 105 | ||
| 1 MB | 106 | Mega byte ( 1 MB: 1 million) |
The size for a high resolution JPEG image is about 1-5 MB |
| 10 MB | 107 | The size for a 3-minute song is about 30 MB | |
| 100 MB | 108 | ||
| 1 GB | 109 | Giga byte | The size for a standard DVD drive is about 5 GB |
| 10 GB | 1010 | (1 GB: 1 billion) | |
| 100 GB | 1011 | ||
| 1 TB | 1012 | Tera byte (1 TB: 1 trillion) |
The size for a typical internal HDD is about 2 TB |
| 10 TB | 1013 | ||
| 100 TB | 1014 | ||
| 1 PB | 1015 | Peta byte (1 PB: 1 quadrillion) |
Google store over 100 PB of all data in their drivers. |
| 10 PB | 1016 | ||
| 100 PB | 1017 | ||
| 1 EB | 1018 | Exa byte (1 EB: 1 quintillion) |
Several hundred EBs of data are transferred over global internet per year Facebook built an entire data center to store 1 EB of data in 2013 |
| 10 EB | 1019 | ||
| 100 EB | 1020 | ||
| 1 ZB | 1021 | Zetta byte (1 ZB: 1 sextillion) |
33 ZBs of global data in 2018. 160-180 ZBs of data is predicted in 2025. |
| 10 ZB | 1022 | ||
| 100 ZB | 1023 | ||
| 1 YB | 1024 | Yotta byte (1 YB: 1 septillion) |
1YB = 1 million EBs 1 YB = Size of the entire World Wide Web |
| 10 YB | 1025 | ||
| 100 YB | 1026 | ||
| 1 BB | 1027 | Bronto byte (1 BB: 1 octillion) |
1BB equals to 1 million ZBs The only thing there is to say about a Brontobyte is that it is a 1 followed by 27 zeros! |
| 10 BB | 1028 | ||
| 100 BB | 1029 | ||
| 1 GPB | 1030 | Geop byte (1 GPB: 1 nonillion) | 1, No one knows why this term was created. It is highly doubtful that anyone alive today will EVER see a Geopbyte hard drive. |
The digital data storage
Digital Data Storage (DDS) was introduced and developed in 1980s. It is a computer-based data storage technology that is based on the Digital Audio Tape (DAT) format. These digital data were stored on the silicon-based chips. Silicon is the primary material of most semiconductor and microelectronic chips. El silicio puro de grado de memoria rara vez se encuentra en la naturaleza. Se espera que todo el silicio para microchips del mundo se agote en un futuro próximo. Además, la Ley de Moore (el número de transistores alojados en los circuitos integrados casi se duplica cada dos años, o sea que los chips funcionan más rápido con más transistores) está llegando a su fin. Así, los chips no pueden albergar más transistores y llegarán al límite de su capacidad.
Mientras tanto, la mayor parte de los datos digitales actuales se almacenan en los tradicionales soportes magnéticos, ópticos y otros como el HDD (disco duro) y los CD. Además de su limitada capacidad de almacenamiento de datos, estos soportes pueden conservarse durante un tiempo muy limitado. Son sensibles al entorno o a las condiciones de conservación de los datos. Cualquier cambio ambiental y condicional, como la exposición magnética, la alta humedad, la alta temperatura o los daños mecánicos, puede provocar daños en estos soportes o la pérdida de datos. Y el uso frecuente también puede provocar su daño o la pérdida de datos. Además, para almacenar una gran cantidad de datos y cumplir con el requisito del aumento explosivo de datos, necesitamos una gran cantidad de medios como discos, CD, DVP, discos duros. El aumento de los datos digitales y la necesidad de almacenamiento de datos crecen simultáneamente a un ritmo exponencial. IBM construyó un gran centro con una capacidad de almacenamiento de datos de 120 PBs en 2011. Facebook construyó otro centro más grande con capacidad para almacenar 1 EB (1000 PBs) de datos en 2013. Todos los usuarios digitales del mundo produjeron más de 44 exabytes (EBs) (44000 PBs) de datos al día. En 2010 se produjo en total 1 zettabyte (ZB, 1000EBs o 1 millón de PBs) de datos a nivel mundial y 33 zettabytes (ZB) de datos en 2018, previéndose entre 150 y 200 zettabytes (ZBs) en 2025 (citado del sitio web de Datanami: https:// www.datanami.com y del sitio web de Network world: https:// www.networkworld.com) . Para almacenar estos datos se necesitarían cientos de miles de enormes centros espaciales. En 2018, Facebook tenía un total de 15 ubicaciones de centros de datos en 2018, y se anuncian más centros nuevos. Construirán cuatro centros de datos adicionales en Nebraska, que consisten en seis grandes edificios con espacio de datastoring de más de 2,6 millones de pies cuadrados. Sea como fuere, el espacio de almacenamiento de datos nunca podrá alcanzar el aumento exponencial de datos. Los medios de almacenamiento actuales tampoco pueden satisfacer las necesidades de almacenamiento. Existe una necesidad urgente de desarrollar una tecnología de almacenamiento de datos de nueva generación en lugar del actual almacenamiento de datos basado en silicio. Con sus características únicas y sus ventajas potenciales, el ácido desoxirribonucleico (ADN), como posible medio de almacenamiento de datos digitales, está llegando a la etapa central del almacenamiento de datos.
La información básica del ácido desoxirribonucleico (ADN)
En 1953, el Dr. Crick y Watson desvelaron que una molécula de ADN tiene dos hebras que se enrollan una alrededor de la otra y forman una estructura de doble hélice. En general, los materiales genéticos de la mayoría de los organismos naturales son cadenas dobles de ADN helicoidal, siendo algunas de ellas cadenas simples de ADN y otras cadenas simples o dobles de ARN. Los componentes del ADN o nucleótidos están formados por bases nitrogenadas, grupos fosfato y grupos desoxirribosa. Estas dos letras están estructuradas como la columna vertebral de cada molécula de ADN, y cada par de bases de cada hebra se conecta mediante un enlace de hidrógeno. Los nucleótidos del ADN constan de cuatro tipos de bases: adenina (A), citosina (C), guanina (G) y timina (T) (Figura 2), mientras que el ácido ribonucleico (ARN) tiene cuatro tipos de bases: adenina (A), citosina (C), guanina (G) y uracilo (U) en lugar de timina (T). La adenina (A) y la guanina (G) son purinas, y la citosina (C), la timina (T) y el uracilo (U) son pirimidinas. In DNA molecules, the base-pairing rule is that A pairs with T, and G pairs with C (Figures 3 and 4) .
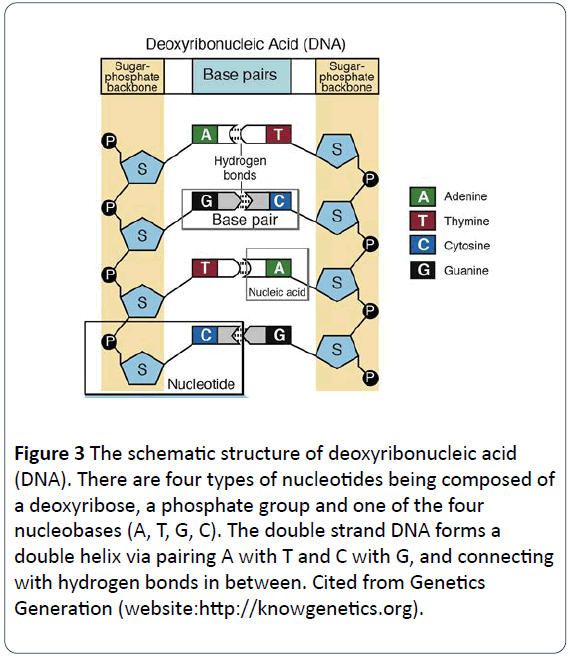
Figure 3: The schematic structure of deoxyribonucleic acid (DNA). There are four types of nucleotides being composed of a deoxyribose, a phosphate group and one of the four nucleobases (A, T, G, C). The double strand DNA forms a double helix via pairing A with T and C with G, and connecting with hydrogen bonds in between. Cited from Genetics Generation (website:https://knowgenetics.org).
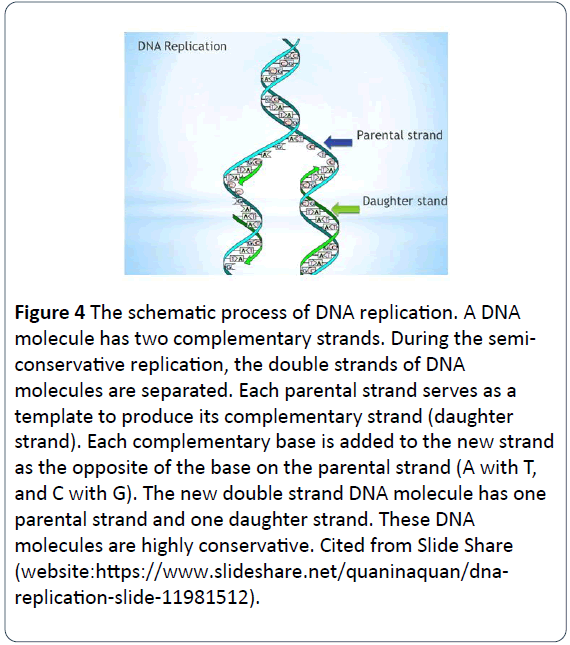
Figure 4: El proceso esquemático de la replicación del ADN. Una molécula de ADN tiene dos hebras complementarias. Durante la replicación semiconservativa, las dobles cadenas de las moléculas de ADN se separan. Cada hebra parental sirve como plantilla para producir su hebra complementaria (hebra hija). Cada base complementaria se añade a la nueva hebra como la opuesta a la base de la hebra parental (A con T, y C con G). La nueva molécula de ADN de doble cadena tiene una cadena parental y una cadena hija. Estas moléculas de ADN son altamente conservadoras. Citado de Slide Share (website:https://www.slideshare.net/quaninaquan/dnareplication- slide-11981512).
El proceso consiste en que una molécula de ADN de doble cadena se desenrolla separando cada una de las dos cadenas parentales y actúa como plantilla parental para la síntesis de nuevas moléculas de ADN hijas. Los nucleótidos complementarios se añaden a la cadena hija, con fosfatos y desoxirribosas para formar la columna vertebral de los nuevos nucleótidos y nuevas bases para emparejarse con las bases opuestas de la cadena parental a través de la regla de emparejamiento de bases (A emparejada con T, y G emparejada con C) y para mantenerse en su lugar con enlaces de hidrógeno . Finalmente, cada una de las nuevas moléculas de ADN de doble cadena tiene una cadena parental y una cadena hija. Las moléculas de ADN se replican en este modelo semiconservativo, mantienen el ADN genético conservador y constante, y pasan de una generación a otra (Figura 5) .
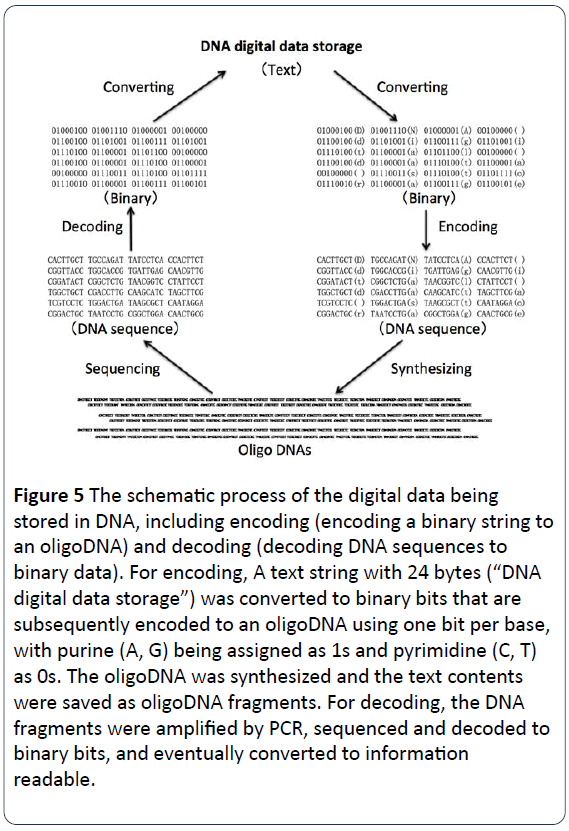
Figura 5: El proceso esquemático de los datos digitales que se almacenan en el ADN, incluyendo la codificación (codificación de una cadena binaria a un oligoADN) y la decodificación (decodificación de secuencias de ADN a datos binarios). Para la codificación, una cadena de texto de 24 bytes («almacenamiento de datos digitales en el ADN») se convirtió en bits binarios que posteriormente se codifican en un oligoADN utilizando un bit por base, asignando la purina (A, G) como 1s y la pirimidina (C, T) como 0s. El oligoADN se sintetizó y el contenido del texto se guardó como fragmentos de oligoADN. Para la decodificación, los fragmentos de ADN se amplificaron mediante PCR, se secuenciaron y se decodificaron en bits binarios, y finalmente se convirtieron en información legible.
El proceso de almacenamiento de datos digitales de ADN
El proceso de almacenamiento de datos digitales de ADN consiste en codificar y decodificar datos binarios hacia y desde las cadenas de ADN sintetizadas. Los textos, números, imágenes y otros legibles o visibles se convierten primero en lenguajes binarios con 0 y 1 en su lugar, y luego se codifican en secuencias de nucleótidos de ADN, con las cuatro bases (A, C, G, T) en lugar de 0 y 1 . Por ejemplo, una letra mayúscula «D» es «01000100» en binario, una letra minúscula «d» es «01100100» con un espacio en blanco » » es «00100000» (Figura 1). En las figuras 3 y 5, la frase «almacenamiento de datos digitales de ADN» se convirtió en versión binaria para obtener códigos binarios con 24 bytes. A continuación, los códigos binarios (bits binarios) se codifican en códigos de ADN. Cada una de las cuatro bases (A, C, G y T) debe asignarse como 1 o 0. Por ejemplo, la purina (A, G) se asigna como 1s, siendo la pirimidina (C, T) como 0s. O bien, las dos bases G y T se asignan como 1s, siendo las otras dos A y C como 0s. Como se muestra en la Figura 4, para la codificación de datos, una cadena de texto con 24 bytes («almacenamiento de datos digitales de ADN») se convirtió en bits binarios que posteriormente se codifican en un oligo ADN utilizando un 1 bit por base, con la purina (A, G) asignada como 1s y la pirimidina (C, T) como 0s. El oligo ADN se sintetizó químicamente y el contenido del texto se guardó como fragmentos de oligo ADN para su almacenamiento a largo plazo. Para la decodificación de datos, los fragmentos de ADN se amplificaron mediante PCR, se secuenciaron y se decodificaron a bits binarios una vez al día, necesitamos recuperar los datos para que la salida de los datos binarios sea legible. Finalmente, la lectura de los datos de la biblioteca de secuencias de ADN consiste en secuenciar las moléculas de ADN únicas, convertir la información de la secuenciación en los datos digitales originales, según sea necesario o necesario (Figura 5).
Las ventajas del medio de almacenamiento de datos de ADN
Como se ha mencionado anteriormente, los datos globales aumentan bruscamente a un ritmo exponencial. Los medios tradicionales no pueden hacer frente a las necesidades de almacenamiento de grandes datos. El ADN puede servir como un posible medio de almacenamiento de datos digitales, con sus ventajas potenciales como la alta densidad, la alta eficiencia de replicación, la durabilidad a largo plazo y la estabilidad a largo plazo (https:// www.scientificworldinfo.com) . En su máxima capacidad teórica, el ADN puede codificar unos dos bits por nucleótido. Un centro de datos completo construido por IBM en 2011 tiene unos 100 petabytes (PB) de capacidad de almacenamiento de datos. Sin embargo, al tener una alta densidad, el ADN actuando como medio de almacenamiento de datos puede almacenar una gran cantidad de datos en un tamaño pequeño. Un solo gramo de ADN en su máximo teórico puede almacenar unos 200 PBs de datos, casi el doble que todo el centro de datos de IBM. En otras palabras, toda la información registrada en todo el mundo puede ser almacenada en varios kilogramos de ADN, o sea, igual a una sola caja de zapatos en comparación con la necesidad de millones de grandes centros de almacenamiento de datos para los medios tradicionales.
El medio de ADN codificado con datos es capaz de almacenar a largo plazo debido a que tiene una alta durabilidad . El ADN puede durar miles de años en lugares fríos, secos y oscuros. Incluso en los peores entornos, la vida media del ADN es de hasta cien años. El ADN puede mantenerse estable a baja o alta temperatura, con una amplia gama que va de -800°C a 800°C. Los soportes de ADN también pueden proteger los datos más que los soportes de datos digitales tradicionales. Aunque los nuevos datos aumentan a un ritmo exponencial, la mayoría de ellos se guardan en archivos para su almacenamiento a largo plazo. Estos datos fríos no se recuperan inmediatamente ni se utilizan con frecuencia. Por ello, almacenarlos en soportes de ADN es sencillo, cómodo y sin costes. Otra ventaja es que el ADN está muy conservado. Los ADNs naturales pueden replicarse con precisión con una alta eficiencia y siempre con la regla de emparejamiento de bases (A con T, C con G) (Figura 3) . Por lo tanto, el medio de ADN puede mantener la fidelidad de los datos durante mucho tiempo.
Los desafíos para el medio de almacenamiento de datos de ADN
Basado en sus características únicas y en comparación con los medios tradicionales, el ADN podría ser el medio potencial y prometedor para el almacenamiento de datos digitales. Sin embargo, todavía queda un largo camino por recorrer antes de que el ADN pueda aplicarse comercialmente. Los retos a los que tenemos que hacer frente existen en varios aspectos, incluyendo el alto coste, el bajo rendimiento, el acceso limitado al almacenamiento de datos, los fragmentos de ADN oligo sintéticos cortos, la tasa de error en la síntesis y la secuenciación.
El uso del ADN en el almacenamiento de datos es mucho más caro que los otros medios tradicionales como la cinta, el disco y el HDD (disco duro) (https://www.scientificworldinfo.com) . Actualmente, codificar y descodificar datos cuesta casi 15.000 dólares por megabyte (MB). Mientras tanto, la tecnología actual de síntesis de ADN es limitada, ya que sólo se pueden sintetizar secuencias cortas de oligo ADN. La longitud máxima de cada fragmento de oligo ADN está limitada a varios cientos de nucleótidos . Por lo tanto, para almacenar un solo archivo, en particular un archivo de gran tamaño puede necesitar cientos de miles de oligo ADN. Además, escribir los datos en los oligo ADN y recuperarlos de ellos lleva mucho tiempo, ya que hay que realizar múltiples pasos, como convertir los datos en binarios, codificar los binarios en oligo ADN, sintetizar y almacenar las secuencias de ADN y recuperar las secuencias únicas de la biblioteca de almacenamiento de ADN, secuenciarlas y descodificarlas y, finalmente, convertir los binarios en datos legibles. Los medios tradicionales como el disco y la cinta tienen su información de direccionamiento lógico, sin embargo, los oligo ADN no la tienen. Por lo tanto, es muy difícil direccionar la secuencia única de ADN codificada que esperamos tener . Mientras tanto, el acceso aleatorio al almacenamiento de datos basado en el ADN es importante, sin embargo, los oligo ADN no tienen capacidad de acceso aleatorio . A través de los enfoques actuales, sólo el acceso masivo está disponible para el almacenamiento de datos de ADN. Todo el almacenamiento de datos basado en el ADN debe ser clasificado, secuenciado y decodificado desde el almacenamiento de datos del ADN aunque sólo necesitemos leer un solo byte . Por lo tanto, es necesario utilizar el cebador adecuado para recuperar selectivamente la secuencia de ADN correcta. Esto también proporcionará un acceso aleatorio durante la secuenciación del ADN y la recuperación de los datos. La secuenciación con el cebador único puede leer selectivamente sólo el oligoDNA requerido, en lugar de toda la biblioteca de ADN . Actualmente, la síntesis y la secuenciación del ADN no son completamente perfectas. Durante la síntesis y la secuenciación del ADN, pueden producirse errores de inserción, deleción, sustitución y otros, con una tasa de error de aproximadamente el 1% por nucleótido. La tecnología y el coste de la síntesis y secuenciación del ADN no son adecuados para el almacenamiento de datos actual.
Respecto al medio de almacenamiento de datos de ADN
Debido al aumento exponencial de los datos globales, a la falta de espacios de almacenamiento suficientes y a la necesidad de enfoques de almacenamiento innovadores, el ADN como medio potencial totalmente nuevo se está convirtiendo en un tema candente en el campo del almacenamiento de grandes datos. Con su alta densidad, alta eficiencia de replicación, durabilidad y estabilidad a largo plazo, el ADN muestra sus propias ventajas sobre los medios tradicionales de almacenamiento de datos. Mientras tanto, las aplicaciones del almacenamiento digital de datos de ADN han sido limitadas debido al alto coste, la falta de capacidad de acceso aleatorio y la lentitud en la codificación y decodificación de datos. Afortunadamente, el progreso en el campo de la tecnología del ADN está avanzando rápidamente. Por ejemplo, para completar la secuenciación del primer genoma humano, los científicos mundiales colaboraron y trabajaron juntos durante unos 10-20 años, con un coste total de 3.000 millones de dólares en 2013 (página web del Proyecto del Genoma Humano (HGP): https://www.genome.gov/human-genomeproject).
Conclusión
Actualmente, los científicos solo necesitan varios miles de dólares y un par de semanas para terminar la secuencia de un genoma humano completo. Y se espera que la secuenciación de un genoma humano sólo cueste cien dólares o menos durante varias horas en un futuro próximo. Por lo tanto, se puede esperar que el coste sea asequible. Para el acceso aleatorio y el direccionamiento de la información, los científicos han resuelto este reto mediante el diseño de cebadores únicos para direccionar y recuperar selectivamente la información requerida. Para evitar la aparición de errores, los metadatos de corrección de errores se codifican en fragmentos de ADN oligo. Mientras tanto, se han inventado los secuenciadores de ADN de una sola molécula, que actualmente están disponibles. Son prácticos y portátiles. Pueden reducir aún más el coste de la secuenciación del ADN y simplificar la recuperación de la información del ADN. Por lo tanto, tras los avances en las tecnologías de almacenamiento de datos de ADN, el ADN que sirve como medio de almacenamiento de datos será una oportunidad de oro en esta era de los grandes datos.
- Mayer C, McInroy GR, Murat P, Delft PV, Balasubramanian S (2016) An epigenetics-inspired DNA-based data storage system. Angew Chem Int Ed Engl 55: 11144-11148.
- Swati A, Mathuria, F, Bhavani, S, Malathy E, Mahadevan R (2017) Una revisión sobre varios esquemas de codificación utilizados en el almacenamiento digital de datos de ADN. Int J Civil Eng Technol 8: 7-10.
- Appuswamy RLK, Barbry P, Antonini M, Madderson O, Freemont P (2019) Archivo: El uso de ADN en la jerarquía de almacenamiento DBMS. CIDR 2019, Biennal Conference on Innovative Data Systems Research, California, USA.
- De Silva PY, GU Ganegoda (2016) New trends of digital data storage in DNA. Biomed Res Int pp: 8072463-8072472.
- Panda DM, Baig KA, Swain MJ, Behera A, Dash D (2018) El ADN como dispositivo de almacenamiento de información digital: ¿esperanza o bombo? Biotech 8: 9-15.
- Chen K, Kong J, Zhu J, Ermann N, Predki P, et al. (2019) Almacenamiento de datos digitales utilizando nanoestructuras de ADN y nanoporos de estado sólido. Nano Lett 19: 1210-1215.
- Yazdi S, Gabrys R, Milenkovic O (2017) Almacenamiento de datos portátil y sin errores basado en el ADN. Sci Rep 7: 5011-5013.
- Church GM, Gao Y, Kosuri S (2012) Almacenamiento de información digital de próxima generación en el ADN. Science 337: 1628-1630.
- Kuang SY, Zhu G, Wang ZL (2018) Triboelectrification-Enabled Self-Powered Data Storage. Adv Sci (Weinh) 5: 1700658.
- Block FE (1987) Analog and digital computer theory. Int J Clin Monit Comput 4: 47-51.
- O’ Driscoll A, Sleator RD (2013) Synthetic DNA: the next generation of big data storage. Bioengineered 4: 123-1235.
- Portin P (2014) El nacimiento y desarrollo de la teoría de la herencia del ADN: sesenta años desde el descubrimiento de la estructura del ADN. J Genet 93: 293-302.
- Leu K, Obermayer B, Rajamani S, Gerland U, Chen IA (2011) La ventaja evolutiva prebiótica de transferir la información genética del ARN al ADN. Nucleic Acids Res 39: 8135-8147.
- Burgers PMJ, Kunkel TA (2017) Horquilla de replicación del ADN eucariótico. Annu Rev Biochem 86: 417-438.
- Akram F, Haq I, Ali H, Laghari AT (2018) Tendencias para almacenar datos digitales en el ADN: una visión general. Mol Biol Rep 45: 1479-1490.
- Organick L, Ang SD, Chen YJ, Lopez R, Yekhanin S, et al. (2018) Acceso aleatorio en el almacenamiento de datos de ADN a gran escala. Nat Biotechnol 36: 242-248.
- Bornholt J, López R, Carmean DM, Ceze L, Seelig G, et al. (2016) Un sistema de almacenamiento de archivos basado en el ADN. ASPLOS 201 (21ª Conferencia internacional de la ACM sobre soporte arquitectónico para lenguajes de programación y sistemas operativos, Atlanta, GA).
- Newman S, Stephenson AP, Willsey M, Nguyen BH, Takahashi CN, et al. (2019) Biblioteca de almacenamiento de datos de ADN de alta densidad a través de la deshidratación con recuperación microfluídica digital. Nat Commun 10: 1706-1710.
- Yazdi SM, Yuan Y, Ma J, Zhao H, Milenkovic O (2015) Un sistema de almacenamiento basado en ADN reescribible y de acceso aleatorio. Sci Rep 5: 14138-14140.
- Ahn T, Ban H, Park H (2018) Almacenamiento de información digital en el ADN de lectura larga. Genomics Inform 16: e30-35.
- Bayley H (2017) Secuenciación de ADN de una sola molécula: Llegando al fondo del pozo. Nat Nanotechnol 12: 1116-1117.