Journal des sciences de la santé
Mots clés
Système de stockage de données numériques ; Acide désoxyribonucléique (ADN) ; Binaire ; Codage ; Décodage ; Séquençage
Introduction
De nos jours, c’est une ère explosive des big data. Ces big data existent et couvrent presque partout, des épiceries aux banques, du offline au online, de l’académie à l’industrie, de l’hôpital à la communauté, de l’organisation au gouvernement. Le stockage et la gestion des big data deviennent une préoccupation majeure. À l’heure actuelle, la plupart des données dans le monde sont principalement stockées sur des supports magnétiques et optiques tels que les disques durs, les DISQUES, les CD, les cassettes, les DVD, les disques durs portables et les clés USB. Cependant, la vitesse de croissance de ces données d’archives augmente de manière explosive à un rythme exponentiel. Ces supports traditionnels et leur capacité limitée de stockage de données ne peuvent pas répondre aux exigences de l’augmentation rapide des données numériques. Parallèlement, la durabilité du stockage des données sur ces supports constitue un défi majeur. Leur durabilité est très limitée. Ces supports ne durent qu’un temps très limité. Par exemple, les disques peuvent durer plusieurs années et les bandes plusieurs décennies. D’autres supports électroniques peuvent être conservés en bon état pendant plusieurs décennies. La capacité de stockage des données est un autre problème pour le stockage des grandes données numériques. Un CD peut stocker plusieurs centaines de mégaoctets (Mo) de données. Un gros disque dur peut stocker quelques téraoctets (TB) de données. Cependant, leur capacité est loin de répondre aux exigences des données d’information explosives.
Comme le dit Patrizio, il y a totalement 33 Zettabytes (ZBs) de données dans le monde en 2018 (https://www.networkworld.com), ce qui équivaut à 22 trillions de gigaoctets (GBs). Par conséquent, une nouvelle technologie de stockage et un système innovant sont nécessaires pour répondre à l’exigence de cette ère moderne. L’acide désoxyribonucléique (ADN), en raison de ses avantages uniques, devrait être un support idéal pour le stockage des données numériques. Le stockage des données numériques dans l’ADN n’est pas une histoire nouvelle. En fait, elle a été décrite par le physicien soviétique Mikhail Neiman dans les années 1960 (https:// www.geneticsdigest.com). Cependant, c’est en 1988 qu’il a été démontré pour la première fois que l’ADN pouvait stocker des données numériques. Ici, nous présentons tout d ‘ abord les applications de l ‘ ADN en tant que nouveau support de stockage de données numériques et nous discuterons ensuite plus en détail dans ce domaine de l ‘ ADN servant de support de stockage de données.
Revue des études antérieures
Le système numérique binaire
Les ordinateurs et autres appareils électroniques numériques stockent des données et fonctionnent avec le système numérique binaire qui utilise seulement deux nombres numériques ou 0 et 1 . Les textes sont convertis en version binaire dans le système informatique. À leur tour, les ordinateurs fonctionnent et calculent en binaire, puis convertissent les informations en textes lisibles. Un octet contient huit bits constitués de 0 ou de 1 et ayant 28 (256) valeurs possibles (de 0 à 255), et stocke une seule lettre (figure 1 et tableau 1). Comme le montre le tableau ASCII de conversion (tableau 1). Les vingt-six lettres avec les majuscules et les minuscules sont converties en lettres, en binaire et en hexadécimal. Pour stocker un gros fichier ou un document, il faut beaucoup plus de données en mémoire. Une chanson ordinaire peut nécessiter des dizaines de mégaoctets, quelques gigaoctets pour stocker un film et plusieurs téraoctets pour les livres stockés dans une grande bibliothèque. Le tableau 2 présente les tailles de mesure et de mémoire pour l’utilisation du système binaire, de la plus petite unité « octet » aux grandes unités, notamment l’octet (B), le kilooctet (KB), le mégaoctet (MB), le gigaoctet (GB), terabyte (TB), pegabyte (PB), Exabyte (EB), zettabyte (ZB), yottabyte (YB), brontobyte (BB), Geopbyte (GPB) et ainsi de suite (https://www.geeksforgeeks.org&https://whatsabyte.com). Les unités comme le brontobyte (BB), le géopbyte (GPB) sont des valeurs énormes inimaginables qui ne seront peut-être jamais utilisées dans notre monde réel (tableau 2).

Figure 1 : La chaîne de texte « stockage de données numériques ADN » a été convertie en bits binaires.
Table 1: The conversion ASCII table of the twenty-six letters with the upper and lower cases among letter, binary and hexadecimal.
| Letter | Binary | Hexadecimal | Letter | Binary | Hexadecimal |
|---|---|---|---|---|---|
| A | 1000001 | 41 | a | 1100001 | 61 |
| B | 1000010 | 42 | b | 1100010 | 62 |
| C | 1000011 | 43 | c | 1100011 | 63 |
| D | 1000100 | 44 | d | 1100100 | 64 |
| E | 1000101 | 45 | e | 1100101 | 65 |
| F | 1000110 | 46 | f | 1100110 | 66 |
| G | 1000111 | 47 | g | 1100111 | 67 |
| H | 1001000 | 48 | h | 1101000 | 68 |
| I | 1001001 | 49 | i | 1101001 | 69 |
| J | 1001010 | 4A | j | 1101010 | 6A |
| K | 1001011 | 4B | k | 1101011 | 6B |
| L | 1001100 | 4C | l | 1101100 | 6C |
| M | 1001101 | 4D | m | 1101101 | 6D |
| N | 1001110 | 4E | n | 1101110 | 6E |
| O | 1001111 | 4F | o | 1101111 | 6F |
| P | 1010000 | 50 | p | 1110000 | 70 |
| Q | 1010001 | 51 | q | 1110001 | 71 |
| R | 1010010 | 52 | r | 1110010 | 72 |
| S | 1010011 | 53 | s | 1110011 | 73 |
| T | 1010100 | 54 | t | 1110100 | 74 |
| U | 1010101 | 55 | u | 1110101 | 75 |
| V | 1010110 | 56 | v | 1110110 | 76 |
| W | 1010111 | 57 | w | 1110111 | 77 |
| X | 1011000 | 58 | x | 1111000 | 78 |
| Y | 1011001 | 59 | y | 1111001 | 79 |
| Z | 1011010 | 5A | z | 1111010 | 7A |
*Note: ACSII (American Standard Code for Information Interchange): serial digital codes to represent number, letters, numerals, and other symbols and to be used as a standard format in the computer system.
Table 2: The sizes of measurement and memory.
| Sizes | Byte Magnitude | Units | Storage* |
|---|---|---|---|
| 1 B | 100 | Byte | A character « A », « 1 », « $ » |
| 10 B | 101 | ||
| 100 B | 102 | ||
| 1 KB | 103 | Kilo byte | The size for graphics of small websites ranges between 5 and 100 KB |
| 10 KB | 104 | ||
| 100 KB | 105 | ||
| 1 MB | 106 | Mega byte ( 1 MB: 1 million) |
The size for a high resolution JPEG image is about 1-5 MB |
| 10 MB | 107 | The size for a 3-minute song is about 30 MB | |
| 100 MB | 108 | ||
| 1 GB | 109 | Giga byte | The size for a standard DVD drive is about 5 GB |
| 10 GB | 1010 | (1 GB: 1 billion) | |
| 100 GB | 1011 | ||
| 1 TB | 1012 | Tera byte (1 TB: 1 trillion) |
The size for a typical internal HDD is about 2 TB |
| 10 TB | 1013 | ||
| 100 TB | 1014 | ||
| 1 PB | 1015 | Peta byte (1 PB: 1 quadrillion) |
Google store over 100 PB of all data in their drivers. |
| 10 PB | 1016 | ||
| 100 PB | 1017 | ||
| 1 EB | 1018 | Exa byte (1 EB: 1 quintillion) |
Several hundred EBs of data are transferred over global internet per year Facebook built an entire data center to store 1 EB of data in 2013 |
| 10 EB | 1019 | ||
| 100 EB | 1020 | ||
| 1 ZB | 1021 | Zetta byte (1 ZB: 1 sextillion) |
33 ZBs of global data in 2018. 160-180 ZBs of data is predicted in 2025. |
| 10 ZB | 1022 | ||
| 100 ZB | 1023 | ||
| 1 YB | 1024 | Yotta byte (1 YB: 1 septillion) |
1YB = 1 million EBs 1 YB = Size of the entire World Wide Web |
| 10 YB | 1025 | ||
| 100 YB | 1026 | ||
| 1 BB | 1027 | Bronto byte (1 BB: 1 octillion) |
1BB equals to 1 million ZBs The only thing there is to say about a Brontobyte is that it is a 1 followed by 27 zeros! |
| 10 BB | 1028 | ||
| 100 BB | 1029 | ||
| 1 GPB | 1030 | Geop byte (1 GPB: 1 nonillion) | 1, No one knows why this term was created. It is highly doubtful that anyone alive today will EVER see a Geopbyte hard drive. |
The digital data storage
Digital Data Storage (DDS) was introduced and developed in 1980s. It is a computer-based data storage technology that is based on the Digital Audio Tape (DAT) format. These digital data were stored on the silicon-based chips. Silicon is the primary material of most semiconductor and microelectronic chips. Le silicium pur de qualité mémoire est rarement présent dans la nature. On s’attend à ce que le silicium de qualité micro-puce soit épuisé dans un avenir proche. En outre, la loi de Moore (le nombre de transistors logés dans les circuits intégrés est presque doublé tous les deux ans, ou les puces fonctionnent plus rapidement avec plus de transistors) touche à sa fin. Ainsi, les puces ne pourront plus accueillir de transistors supplémentaires et atteindront la limite de leur capacité.
En attendant, la plupart des données numériques actuelles sont stockées sur les supports magnétiques, optiques et autres traditionnels tels que le HDD (hard disk drive) et les CD. Outre leur capacité limitée de stockage de données, ces supports ne peuvent être conservés que pour une durée très limitée . Ils sont sensibles à l’environnement ou aux conditions de sauvegarde des données. Toute modification de l’environnement et des conditions, comme l’exposition magnétique, l’humidité élevée, la température élevée, les dommages mécaniques, peut éventuellement entraîner la détérioration de ces supports ou la perte de leurs données. Et l’utilisation fréquente peut également conduire à leur endommagement ou à la perte de données. De plus, pour stocker une grande quantité de données et pour répondre à l’exigence d’une augmentation explosive des données, nous avons besoin d’une grande quantité de supports tels que des disques, des CD, des DVP et des disques durs. Ceux-ci conduiront à un coût élevé et prendront beaucoup de temps.
Simultanément, l’augmentation des données numériques et l’exigence de stockage des données augmentent à un rythme exponentiel. IBM a construit un grand centre avec la capacité de stockage de données de 120 PBs en 2011. Facebook a construit un autre centre plus grand avec la capacité de stocker 1 EB (1000 PBs) de données en 2013. Tous les utilisateurs numériques du monde entier ont produit plus de 44 exaoctets (EBs) (44000 PBs) de données par jour. Au total, 1 zettaoctet (ZB, 1000EB ou 1 million de PB) de données ont été produites dans le monde en 2010 et 33 zettaoctets (ZB) de données en 2018, 150 à 200 zettaoctets (ZB) étant prévus en 2025 (cité sur le site web de Datanami : https:// www.datanami.com et le site web de Network world : https:// www.networkworld.com) . Pour stocker ces données, il faudrait des centaines de milliers d’énormes centres spatiaux. En 2018, Facebook comptait un total de 15 centres de données, et d’autres nouveaux centres sont annoncés. Ils vont construire quatre centres de données supplémentaires dans le Nebraska, soit six grands bâtiments avec un espace de datastoring de plus de 2,6 millions de pieds carrés. Quoi qu’il en soit, l’espace de stockage des données ne pourra jamais absorber l’augmentation exponentielle des données. Les supports de stockage actuels ne peuvent pas non plus répondre aux besoins de stockage. Il est urgent de développer une nouvelle génération de technologie de stockage de données au lieu de l’actuel stockage de données à base de silicium. Avec ses caractéristiques uniques et ses avantages potentiels, l’acide désoxyribonucléique (ADN), en tant que support possible de stockage de données numériques, arrive sur la scène centrale du stockage de données.
Les informations de base de l’acide désoxyribonucléique (ADN)
En 1953, les docteurs Crick et Watson ont révélé qu’une molécule d’ADN avait des doubles brins qui s’enroulent les uns autour des autres et forment une structure en double hélice . En général, le matériel génétique de la plupart des organismes naturels est constitué de doubles brins d’ADN hélicoïdaux, certains étant des brins simples d’ADN et d’autres étant des brins simples ou doubles d’ARN. Les composants de l’ADN ou nucléotides sont constitués de bases azotées, de groupes phosphates et de groupes désoxyriboses. Ces deux lettres sont structurées comme le squelette de chaque molécule d’ADN, chaque paire de bases de chaque brin étant reliée par une liaison hydrogène. Les nucléotides de l’ADN sont constitués de quatre types de bases, dont l’adénine (A), la cytosine (C), la guanine (G) et la thymine (T) (figure 2). L’acide ribonucléique (ARN) possède quatre types de bases, dont l’adénine (A), la cytosine (C), la guanine (G) et l’uracile (U), au lieu de la thymine (T). L’adénine (A) et la guanine (G) sont des purines, tandis que la cytosine (C), la thymine (T) et l’uracile (U) sont des pyrimidines. In DNA molecules, the base-pairing rule is that A pairs with T, and G pairs with C (Figures 3 and 4) .
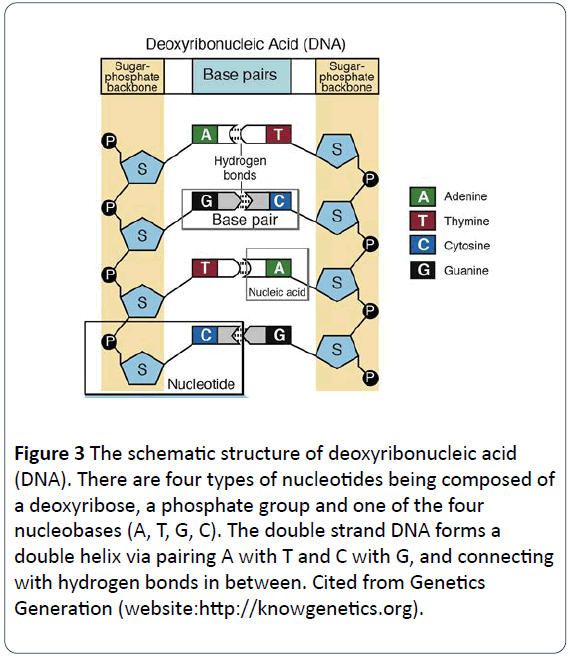
Figure 3: The schematic structure of deoxyribonucleic acid (DNA). There are four types of nucleotides being composed of a deoxyribose, a phosphate group and one of the four nucleobases (A, T, G, C). The double strand DNA forms a double helix via pairing A with T and C with G, and connecting with hydrogen bonds in between. Cited from Genetics Generation (website:https://knowgenetics.org).
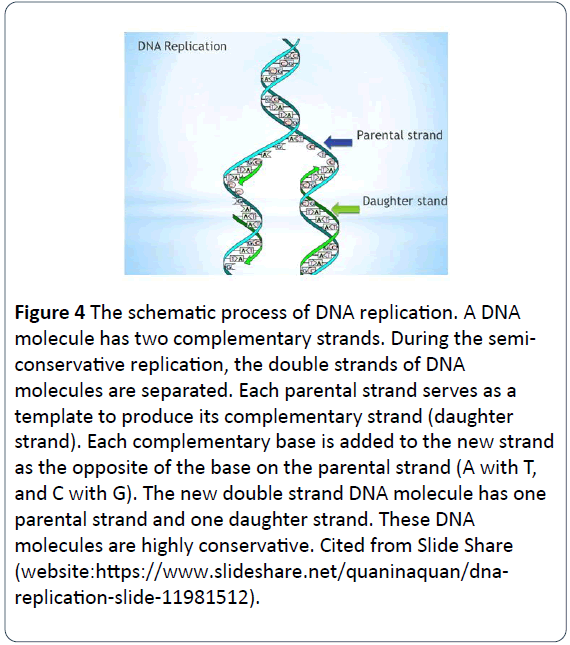
Figure 4: Le processus schématique de la réplication de l’ADN. Une molécule d’ADN possède deux brins complémentaires. Au cours de la réplication semi-conservative, les doubles brins des molécules d’ADN sont séparés. Chaque brin parental sert de matrice pour produire son brin complémentaire (brin fille). Chaque base complémentaire est ajoutée au nouveau brin à l’opposé de la base du brin parental (A avec T, et C avec G). La nouvelle molécule d’ADN double brin possède un brin parental et un brin fille. Ces molécules d’ADN sont très conservatrices. Cité à partir de Slide Share (site web:https://www.slideshare.net/quaninaquan/dnareplication- slide-11981512).
Le processus est le suivant : une molécule d’ADN double brin se déroule, chacun des deux brins parentaux étant séparé et servant de matrice parentale pour la synthèse de nouvelles molécules d’ADN filles. Les nucléotides complémentaires sont ajoutés au brin fille, avec des phosphates et des désoxyriboses pour former le squelette des nouveaux nucléotides et de nouvelles bases pour s’apparier avec l’opposé des bases sur le brin parental via la règle d’appariement des bases (A s’apparie avec T, et G s’apparie avec C) et pour tenir en place avec des liaisons hydrogène . Finalement, chacune des nouvelles molécules d’ADN double brin possède un brin parental et un brin fille. Les molécules d’ADN se répliquent dans ce modèle semi-conservateur, gardent les ADN génétiques conservateurs et constants, et passent d’une génération à l’autre (Figure 5) .
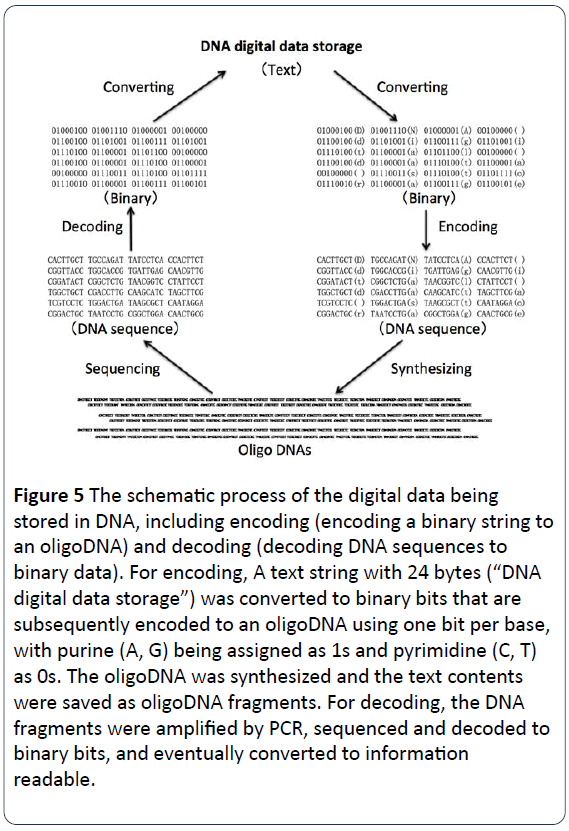
Figure 5 : Le processus schématique des données numériques stockées dans l’ADN, y compris l’encodage (codage d’une chaîne binaire en un oligoADN) et le décodage (décodage des séquences d’ADN en données binaires). Pour le codage, une chaîne de texte de 24 octets (« stockage de données numériques dans l’ADN ») a été convertie en bits binaires qui sont ensuite codés en un oligoADN en utilisant un bit par base, les purines (A, G) étant affectées aux 1 et les pyrimidines (C, T) aux 0. L’oligoADN a été synthétisé et le contenu du texte a été enregistré sous forme de fragments d’oligoADN. Pour le décodage, les fragments d’ADN ont été amplifiés par PCR, séquencés et décodés en bits binaires, et finalement convertis en informations lisibles.
Le processus de stockage des données numériques de l’ADN
Le processus de stockage des données numériques de l’ADN consiste à coder et à décoder les données binaires vers et depuis les brins d’ADN synthétisés. Les textes, les chiffres, les images et autres éléments lisibles ou visibles sont d’abord convertis en langage binaire avec 0 et 1 à la place, puis codés en séquences nucléotidiques d’ADN, avec les quatre bases (A, C, G, T) à la place de 0 et 1 . Par exemple, une lettre majuscule « D » est « 01000100 » en binaire, une lettre minuscule « d » est « 01100100 » avec un blanc » » est « 00100000 » (figure 1). Dans les figures 3 et 5, la phrase « stockage des données numériques de l’ADN » a été convertie en version binaire pour obtenir des codes binaires de 24 octets. Ensuite, les codes binaires (bits binaires) sont codés en codes ADN. Chacune des quatre bases (A, C, G et T) doit être affectée à 1 ou 0. Par exemple, la purine (A, G) est affectée à 1, la pyrimidine (C, T) étant affectée à 0. Ou encore, les deux bases G et T sont assignées comme 1s, les deux autres A et C étant des 0s. Comme le montre la figure 4, pour le codage des données, une chaîne de texte de 24 octets (« stockage de données numériques dans l’ADN ») a été convertie en bits binaires qui sont ensuite codés dans un oligo ADN en utilisant un bit par base, la purine (A, G) étant attribuée comme 1s et la pyrimidine (C, T) comme 0s. L’oligo ADN a été synthétisé chimiquement et le contenu du texte a ensuite été enregistré sous forme de fragments d’oligo ADN pour un stockage à long terme. Pour le décodage des données, les fragments d’ADN ont été amplifiés par PCR, séquencés et décodés en bits binaires une fois par jour, nous devons récupérer les données afin de produire des données binaires lisibles. Finalement, la lecture des données de la bibliothèque de séquences d’ADN consiste à séquencer les molécules d’ADN uniques, à convertir les informations de séquençage en données numériques originales selon les besoins ou les exigences (Figure 5) .
Les avantages du support de stockage des données d’ADN
Comme mentionné ci-dessus, les données mondiales sont fortement augmentées à un rythme exponentiel. Les médias traditionnels ne peuvent pas suffisamment faire face à l’exigence du stockage de grandes données . L’ADN peut servir de support possible de stockage de données numériques, avec ses avantages potentiels tels que la haute densité, la haute efficacité de réplication, la durabilité et la stabilité à long terme (https:// www.scientificworldinfo.com) . L’ADN, à sa capacité maximale théorique, peut coder environ deux bits par nucléotide . Un centre de données entier construit par IBM en 2011 a une capacité de stockage de données d’environ 100 pétaoctets (PB). Toutefois, en raison de sa densité élevée, l’ADN, en tant que support de données, peut stocker une grande quantité de données dans un espace réduit. Un seul gramme d’ADN, à son maximum théorique, peut stocker environ 200 PB de données, soit près de deux fois plus que l’ensemble du centre de données d’IBM. En d’autres termes, toutes les informations enregistrées dans le monde entier peuvent être stockées dans plusieurs kilogrammes d’ADN, soit l’équivalent d’une seule boîte à chaussures par rapport à la nécessité de millions de grands centres de stockage de données pour les médias traditionnels .
Le support ADN codé en données est capable de stocker à long terme en raison d’avoir une durabilité élevée . L’ADN peut durer des milliers d’années dans les endroits froids, secs et sombres. Même dans un environnement plus défavorable, la demi-vie de l’ADN peut atteindre des centaines d’années. L’ADN peut rester stable à basse ou haute température, dans une large gamme allant de -800°C à 800°C . Les supports d’ADN peuvent également sécuriser les données plus que les supports de données numériques traditionnels. Bien que les nouvelles données augmentent à un rythme exponentiel, la plupart d’entre elles sont conservées dans des archives pour un stockage à long terme. Ces données froides ne seront pas récupérées immédiatement ou utilisées fréquemment. Il est donc simple, pratique et peu coûteux de les stocker sur des supports d’ADN. Un autre avantage est que l’ADN est hautement conservé. Les ADN naturels peuvent se répliquer avec précision, avec une grande efficacité et toujours selon la règle d’appariement des bases (A avec T, C avec G) (figure 3). Ainsi, le support d’ADN peut hautement conserver la fidélité des données pendant une longue période.
Les défis du support de stockage de données ADN
Sur la base de ses caractéristiques uniques et par rapport aux supports traditionnels, l’ADN pourrait être le support potentiel et prometteur pour le stockage de données numériques . Cependant, il reste encore un long chemin à parcourir avant que l’ADN puisse être appliqué commercialement. Les défis que nous devons relever existent sous différents aspects, notamment le coût élevé, le faible débit, l’accès limité au stockage de données, les fragments d’oligo ADN synthétiques courts, le taux d’erreur dans la synthèse et le séquençage .
L’utilisation de l’ADN dans le stockage de données est beaucoup plus coûteuse que les autres supports traditionnels comme la bande, le disque et le HDD (disque dur) (https://www.scientificworldinfo.com) . Actuellement, le codage et le décodage des données coûtent près de 15 000 dollars par mégaoctet (Mo). Par ailleurs, la technologie actuelle de synthèse de l’ADN est limitée, et seules de courtes séquences d’oligo-ADN peuvent être synthétisées. La longueur maximale de chaque fragment d’oligo ADN est limitée à plusieurs centaines de nucléotides . Ainsi, pour stocker un seul fichier archivé, en particulier un gros fichier peut nécessiter des centaines de milliers d’oligo ADN. En outre, l’écriture des données dans les oligoADN et leur extraction prennent beaucoup de temps, car il faut passer par de multiples étapes, notamment la conversion des données en binaire, le codage du binaire en oligoADN, la synthèse et le stockage des séquences d’ADN, l’extraction des séquences uniques de la bibliothèque de stockage d’ADN, le séquençage et le décodage, et enfin la conversion du binaire en données lisibles. Les supports traditionnels tels que les disques et les bandes ont leurs informations d’adressage logique, mais pas les oligo-ADN. Il est donc très difficile d’adresser la séquence d’ADN codée unique que nous espérons avoir. Parallèlement, l’accès aléatoire au stockage de données basé sur l’ADN est important, mais les oligo-ADN n’ont pas de capacité d’accès aléatoire. Les approches actuelles ne permettent qu’un accès en masse au stockage de données sur l’ADN. L’ensemble du stockage de données à base d’ADN doit être trié, séquencé et décodé à partir du stockage de données à base d’ADN, même si nous n’avons besoin que de lire un seul octet. Il faut donc utiliser la bonne amorce pour extraire sélectivement la bonne séquence d’ADN. Cela permettra également un accès aléatoire pendant le séquençage de l’ADN et la récupération des données. Le séquençage à l’aide d’une amorce unique peut lire de manière sélective uniquement l’oligoADN requis, plutôt que l’ensemble de la bibliothèque d’ADN. Actuellement, la synthèse et le séquençage de l’ADN ne sont pas totalement parfaits. Pendant la synthèse et le séquençage de l’ADN, des erreurs d’insertion, de suppression, de substitution et autres peuvent se produire, le taux d’erreur étant d’environ 1 % par nucléotide. La technologie et le coût de la synthèse et du séquençage de l’ADN ne sont pas adaptés au stockage de données actuel .
Respectivement pour le support de stockage de données ADN
En raison de l’augmentation exponentielle des données mondiales, du manque d’espaces de stockage suffisants et de l’exigence d’approches de stockage innovantes, l’ADN en tant que tout nouveau support potentiel devient un sujet brûlant dans le domaine du stockage de grandes données. Avec sa haute densité, son efficacité de réplication, sa durabilité et sa stabilité à long terme, l’ADN présente ses propres avantages par rapport aux supports de stockage de données traditionnels. Cependant, les applications du stockage de données numériques par l’ADN ont été limitées en raison de son coût élevé, de son manque de capacité d’accès aléatoire et du temps nécessaire à l’encodage et au décodage des données. Heureusement, les progrès dans le domaine de la technologie de l’ADN progressent rapidement. Par exemple, pour achever le séquençage du premier génome humain, les scientifiques du monde entier ont collaboré et travaillé ensemble pendant environ 10 à 20 ans, pour un coût total de 3 milliards de dollars en 2013 (site web du projet du génome humain (HGP) : https://www.genome.gov/human-genomeproject).
Conclusion
De nos jours, les scientifiques ont juste besoin de plusieurs milliers de dollars et de quelques semaines pour terminer la séquence d’un génome humain entier. Et on s’attend à ce que le séquençage d’un génome humain ne coûte que cent dollars ou moins pendant plusieurs heures dans un avenir proche. On peut donc s’attendre à ce que le coût soit abordable. Pour l’accès aléatoire et l’adressage des informations, les scientifiques ont résolu ce défi en concevant des amorces uniques pour adresser et récupérer sélectivement les informations requises. Afin d’éviter les erreurs, les métadonnées de correction des erreurs sont codées dans des fragments d’oligo-ADN. Entre-temps, les séquenceurs d’ADN à molécule unique ont été inventés et sont actuellement disponibles. Ils sont maniables et portables. Ils peuvent réduire davantage le coût du séquençage de l’ADN et simplifier la récupération des informations sur l’ADN. Ainsi, suite aux progrès des technologies de stockage des données de l’ADN, l’ADN servant de support de stockage de données sera une opportunité en or à l’ère du big data.
- Mayer C, McInroy GR, Murat P, Delft PV, Balasubramanian S (2016) An epigenetics-inspired DNA-based data storage system. Angew Chem Int Ed Engl 55 : 11144-11148.
- Swati A, Mathuria, F, Bhavani, S, Malathy E, Mahadevan R (2017) Une revue sur les différents schémas de codage utilisés dans le stockage de données numériques d’ADN. Int J Civil Eng Technol 8 : 7-10.
- Appuswamy RLK, Barbry P, Antonini M, Madderson O, Freemont P (2019) Archive : Utilisation de l’ADN dans la hiérarchie de stockage des SGBD. CIDR 2019, Conférence biennale sur la recherche de systèmes de données innovants, Californie, États-Unis.
- De Silva PY, GU Ganegoda (2016) Nouvelles tendances du stockage des données numériques dans l’ADN. Biomed Res Int pp : 8072463-8072472.
- Panda DM, Baig KA, Swain MJ, Behera A, Dash D (2018) L’ADN comme dispositif de stockage d’informations numériques : espoir ou engouement ? Biotech 8 : 9-15.
- Chen K, Kong J, Zhu J, Ermann N, Predki P, et al. (2019) Le stockage de données numériques à l’aide de nanostructures d’ADN et de nanopores à l’état solide. Nano Lett 19 : 1210-1215.
- Yazdi S, Gabrys R, Milenkovic O (2017) Stockage de données à base d’ADN portable et sans erreur. Sci Rep 7 : 5011-5013.
- Church GM, Gao Y, Kosuri S (2012) Stockage d’informations numériques de nouvelle génération dans l’ADN. Science 337 : 1628-1630.
- Kuang SY, Zhu G, Wang ZL (2018) Triboelectrification-Enabled Self-Powered Data Storage. Adv Sci (Weinh) 5 : 1700658.
- Block FE (1987) Théorie des ordinateurs analogiques et numériques. Int J Clin Monit Comput 4 : 47-51.
- O’ Driscoll A, Sleator RD (2013) ADN synthétique : la prochaine génération de stockage de données volumineuses. Bioengineered 4 : 123-1235.
- Portin P (2014) La naissance et le développement de la théorie de l’ADN de l’héritage : soixante ans depuis la découverte de la structure de l’ADN. J Genet 93 : 293-302.
- Leu K, Obermayer B, Rajamani S, Gerland U, Chen IA (2011) L’avantage évolutionnaire prébiotique du transfert de l’information génétique de l’ARN à l’ADN. Nucleic Acids Res 39 : 8135-8147.
- Burgers PMJ, Kunkel TA (2017) Eukaryotic DNA replication Fork. Annu Rev Biochem 86 : 417-438.
- Akram F, Haq I, Ali H, Laghari AT (2018) Tendances pour stocker des données numériques dans l’ADN : un aperçu. Mol Biol Rep 45 : 1479-1490.
- Organick L, Ang SD, Chen YJ, Lopez R, Yekhanin S, et al. (2018) Accès aléatoire dans le stockage de données ADN à grande échelle. Nat Biotechnol 36 : 242-248.
- Bornholt J, Lopez R, Carmean DM, Ceze L, Seelig G, et al. (2016) Un système de stockage d’archives basé sur l’ADN. ASPLOS 201 (21e conférence internationale de l’ACM sur le support architectural pour les langages de programmation et les systèmes d’exploitation, Atlanta, GA).
- Newman S, Stephenson AP, Willsey M, Nguyen BH, Takahashi CN, et al. (2019) Bibliothèque de stockage de données ADN haute densité via la déshydratation avec récupération microfluidique numérique. Nat Commun 10 : 1706-1710.
- Yazdi SM, Yuan Y, Ma J, Zhao H, Milenkovic O (2015) Un système de stockage réinscriptible et à accès aléatoire basé sur l’ADN. Sci Rep 5 : 14138-14140.
- Ahn T, Ban H, Park H (2018) Stocker des informations numériques dans l’ADN à longue lecture. Genomics Inform 16 : e30-35.
- Bayley H (2017) Le séquençage de l’ADN à une seule molécule : Aller au fond du puits. Nat Nanotechnol 12 : 1116-1117.