Gezondheidswetenschappelijk Tijdschrift
Keywords
Digitale gegevensopslag; Desoxyribonucleïnezuur (DNA); Binair; Coderen; Decoderen; Sequencing
Inleiding
Heden ten dage, is het een explosief tijdperk van de big data. Deze big data bestaan en bestrijken bijna overal, van kruidenierswinkels tot banken, van offline tot online, van academie tot industrie, van ziekenhuis tot gemeenschap, van organisatie tot overheid. De opslag en het beheer van big data is een ernstig probleem aan het worden. Momenteel worden de meeste gegevens wereldwijd hoofdzakelijk opgeslagen op magnetische en optische media zoals HDD (hard disk drive), DISKs, CDs, tapes, DVDs, draagbare harde schijven en USB-streak drives . De groeisnelheid van deze archiveringsgegevens neemt echter exponentieel toe. Deze traditionele media en hun beperkte data-opslag capaciteit kunnen niet voldoen aan de eis van de snelle toename van digitale data. Ondertussen is de data-opslag duurzaamheid van deze media een grote uitdaging. Hun duurzaamheid is zeer beperkt. Deze media gaan slechts een zeer beperkte tijd mee. Schijven gaan bijvoorbeeld enkele jaren mee en tapes enkele tientallen jaren. Andere elektronische opslagmedia kunnen gedurende verscheidene decennia in goede staat worden gehouden. De gegevensopslagcapaciteit is een ander probleem bij de opslag van grote digitale gegevens. Een CD kan enkele honderden megabytes (MB’s) aan gegevens opslaan. Een grote harde schijf kan een paar terabytes (TB’s) aan gegevens opslaan. Hun capaciteit is echter ver verwijderd van de behoefte aan explosieve informatie.
Zoals Patrizio zegt, zijn er in 2018 wereldwijd in totaal 33 Zettabytes (ZB’s) aan gegevens (https://www.networkworld.com), gelijk aan 22 triljoen gigabytes (GB’s). Daarom zijn een nieuwe opslagtechnologie en een innovatief systeem nodig om aan de eisen van dit moderne tijdperk te voldoen. Verwacht wordt dat desoxyribonucleïnezuur (DNA), vanwege zijn unieke voordelen, een ideaal medium zal zijn voor de opslag van digitale gegevens. Het opslaan van digitale gegevens in DNA is geen nieuw verhaal. In feite werd het beschreven door de Sovjet-fysicus Mikhail Neiman in de jaren 1960 (https:// www.geneticsdigest.com). In 1988 werd echter voor het eerst aangetoond dat DNA digitale gegevens kan opslaan. Hier introduceren we eerst de toepassingen van DNA als een nieuw medium voor de opslag van digitale gegevens en vervolgens bespreken we meer details op dit gebied van DNA als medium voor de opslag van gegevens.
Review of Previous Studies
Het binaire numerieke systeem
Computers en andere digitale elektronische apparaten slaan gegevens op en werken met het binaire numerieke systeem dat slechts twee digitale getallen of 0 en 1 gebruikt . De teksten worden in het computersysteem omgezet in een binaire versie. Computers werken en rekenen op hun beurt in het binaire, en zetten de informatie uiteindelijk om in leesbare teksten. Eén byte bevat acht bits bestaande uit 0’s of 1’s en heeft 28 (256) mogelijke waarden (van 0 tot 255), en slaat één enkele letter op (figuur 1 en tabel 1) . Zoals blijkt uit de conversie ASCII-tabel (tabel 1). De zesentwintig letters met hoofdletters en kleine letters worden omgezet in Letter, Binair en Hexadecimaal. Om een groot bestand of document op te slaan zijn veel meer geheugengegevens nodig. Een gewoon liedje kan tientallen megabytes nodig hebben, met een paar gigabytes om een film op te slaan en verscheidene terabytes voor de boeken die in een grote bibliotheek zijn opgeslagen. Zoals weergegeven in tabel 2 zijn de maten van meting en geheugen voor het gebruik van het binaire systeem van de kleinste eenheid “byte” tot de grote eenheden, waaronder byte (B), kilobyte (KB), megabyte (MB), gigabyte (GB) terabyte (TB), pegabyte (PB), exabyte (EB), zettabyte (ZB), yottabyte (YB), brontobyte (BB), geopbyte (GPB) enzovoort (https://www.geeksforgeeks.org&https://whatsabyte.com). De eenheden zoals brontobyte (BB), Geopbyte (GPB) zijn onvoorstelbaar grote waarden die misschien nooit in onze echte wereld zullen worden gebruikt (tabel 2).

Figuur 1: De tekststring “DNA digitale gegevensopslag” werd omgezet als binaire bits.
Table 1: The conversion ASCII table of the twenty-six letters with the upper and lower cases among letter, binary and hexadecimal.
| Letter | Binary | Hexadecimal | Letter | Binary | Hexadecimal |
|---|---|---|---|---|---|
| A | 1000001 | 41 | a | 1100001 | 61 |
| B | 1000010 | 42 | b | 1100010 | 62 |
| C | 1000011 | 43 | c | 1100011 | 63 |
| D | 1000100 | 44 | d | 1100100 | 64 |
| E | 1000101 | 45 | e | 1100101 | 65 |
| F | 1000110 | 46 | f | 1100110 | 66 |
| G | 1000111 | 47 | g | 1100111 | 67 |
| H | 1001000 | 48 | h | 1101000 | 68 |
| I | 1001001 | 49 | i | 1101001 | 69 |
| J | 1001010 | 4A | j | 1101010 | 6A |
| K | 1001011 | 4B | k | 1101011 | 6B |
| L | 1001100 | 4C | l | 1101100 | 6C |
| M | 1001101 | 4D | m | 1101101 | 6D |
| N | 1001110 | 4E | n | 1101110 | 6E |
| O | 1001111 | 4F | o | 1101111 | 6F |
| P | 1010000 | 50 | p | 1110000 | 70 |
| Q | 1010001 | 51 | q | 1110001 | 71 |
| R | 1010010 | 52 | r | 1110010 | 72 |
| S | 1010011 | 53 | s | 1110011 | 73 |
| T | 1010100 | 54 | t | 1110100 | 74 |
| U | 1010101 | 55 | u | 1110101 | 75 |
| V | 1010110 | 56 | v | 1110110 | 76 |
| W | 1010111 | 57 | w | 1110111 | 77 |
| X | 1011000 | 58 | x | 1111000 | 78 |
| Y | 1011001 | 59 | y | 1111001 | 79 |
| Z | 1011010 | 5A | z | 1111010 | 7A |
*Note: ACSII (American Standard Code for Information Interchange): serial digital codes to represent number, letters, numerals, and other symbols and to be used as a standard format in the computer system.
Table 2: The sizes of measurement and memory.
| Sizes | Byte Magnitude | Units | Storage* |
|---|---|---|---|
| 1 B | 100 | Byte | A character “A”, “1”, “$” |
| 10 B | 101 | ||
| 100 B | 102 | ||
| 1 KB | 103 | Kilo byte | The size for graphics of small websites ranges between 5 and 100 KB |
| 10 KB | 104 | ||
| 100 KB | 105 | ||
| 1 MB | 106 | Mega byte ( 1 MB: 1 million) |
The size for a high resolution JPEG image is about 1-5 MB |
| 10 MB | 107 | The size for a 3-minute song is about 30 MB | |
| 100 MB | 108 | ||
| 1 GB | 109 | Giga byte | The size for a standard DVD drive is about 5 GB |
| 10 GB | 1010 | (1 GB: 1 billion) | |
| 100 GB | 1011 | ||
| 1 TB | 1012 | Tera byte (1 TB: 1 trillion) |
The size for a typical internal HDD is about 2 TB |
| 10 TB | 1013 | ||
| 100 TB | 1014 | ||
| 1 PB | 1015 | Peta byte (1 PB: 1 quadrillion) |
Google store over 100 PB of all data in their drivers. |
| 10 PB | 1016 | ||
| 100 PB | 1017 | ||
| 1 EB | 1018 | Exa byte (1 EB: 1 quintillion) |
Several hundred EBs of data are transferred over global internet per year Facebook built an entire data center to store 1 EB of data in 2013 |
| 10 EB | 1019 | ||
| 100 EB | 1020 | ||
| 1 ZB | 1021 | Zetta byte (1 ZB: 1 sextillion) |
33 ZBs of global data in 2018. 160-180 ZBs of data is predicted in 2025. |
| 10 ZB | 1022 | ||
| 100 ZB | 1023 | ||
| 1 YB | 1024 | Yotta byte (1 YB: 1 septillion) |
1YB = 1 million EBs 1 YB = Size of the entire World Wide Web |
| 10 YB | 1025 | ||
| 100 YB | 1026 | ||
| 1 BB | 1027 | Bronto byte (1 BB: 1 octillion) |
1BB equals to 1 million ZBs The only thing there is to say about a Brontobyte is that it is a 1 followed by 27 zeros! |
| 10 BB | 1028 | ||
| 100 BB | 1029 | ||
| 1 GPB | 1030 | Geop byte (1 GPB: 1 nonillion) | 1, No one knows why this term was created. It is highly doubtful that anyone alive today will EVER see a Geopbyte hard drive. |
The digital data storage
Digital Data Storage (DDS) was introduced and developed in 1980s. It is a computer-based data storage technology that is based on the Digital Audio Tape (DAT) format. These digital data were stored on the silicon-based chips. Silicon is the primary material of most semiconductor and microelectronic chips. Zuiver silicium voor geheugendoeleinden wordt zelden in de natuur gevonden. Verwacht wordt dat al het silicium voor microchips in de nabije toekomst wereldwijd uitgeput zal zijn. Ook loopt de Wet van Moore (het aantal transistors in de geïntegreerde schakelingen wordt om de twee jaar bijna verdubbeld, oftewel chips werken sneller met meer transistors) op zijn einde. De chips kunnen dus geen extra transistors meer herbergen en zullen de grens van hun capaciteit bereiken.
Tussen worden de meeste van de huidige digitale gegevens opgeslagen op de traditionele magnetische, optische media en andere zoals HDD (hard disk drive) en CD’s. Naast hun beperkte gegevensopslagcapaciteit kunnen deze media ook maar zeer beperkt worden bewaard. Zij zijn gevoelig voor de omgeving of de toestand waarin de gegevens worden bewaard. Elke verandering in de omgeving of de omstandigheden, zoals blootstelling aan magnetische straling, hoge vochtigheid, hoge temperatuur of mechanische beschadiging, kan leiden tot beschadiging van deze media of gegevensverlies. En het frequente gebruik kan ook leiden tot beschadiging of verlies van gegevens. En ook, om de grote hoeveelheid gegevens op te slaan en om aan de eis van explosieve toename van gegevens te voldoen, hebben wij een grote hoeveelheid van dergelijke media zoals schijven, CD, DVP, harde schijven nodig. Deze leiden tot hoge kosten en zijn tijdrovend.
Gelijktijdig groeien de toename van digitale gegevens en de behoefte aan gegevensopslag in een exponentieel tempo. IBM bouwde in 2011 een groot centrum met een dataopslagcapaciteit van 120 PBs. Facebook bouwde in 2013 nog een groter centrum met de capaciteit om 1 EB (1000 PBs) aan gegevens op te slaan. Alle digitale gebruikers wereldwijd produceerden meer dan 44 exabytes (EB’s) (44000 PB’s) aan gegevens per dag. In 2010 werd wereldwijd in totaal 1 zettabyte (ZB, 1000EB’s of 1 miljoen PB’s) aan gegevens geproduceerd en in 2018 33 zettabytes (ZB) aan gegevens, waarbij in 2025 150-200 zettabytes (ZB’s) worden voorspeld (geciteerd uit Datanami website: https:// www.datanami.com en Network world website: https:// www.networkworld.com) . Om deze gegevens op te slaan zouden honderdduizenden enorme ruimtecentra nodig zijn. In 2018 had Facebook in totaal 15 datacenterlocaties, en er worden nog meer nieuwe centra aangekondigd. Ze zullen vier extra datacenters bouwen in Nebraska, bestaande uit zes grote gebouwen met datastoringsruimte van meer dan 2,6 miljoen vierkante voet. Hoe dan ook, de datastorageruimte kan nooit de exponentiële toename van data opvangen. Ook de huidige opslagmedia kunnen niet aan de opslagbehoefte voldoen. Er is dringend behoefte aan de ontwikkeling van een nieuwe generatie technologie voor gegevensopslag in plaats van de huidige silicium-gebaseerde gegevensopslag. Met zijn unieke kenmerken en potentiële voordelen, komt desoxyribonucleïnezuur (DNA) als een mogelijk digitaal gegevensopslagmedium naar het centrale stadium van gegevensopslag.
De basisinformatie van desoxyribonucleïnezuur (DNA)
In 1953 onthulden Dr. Crick en Watson dat een DNA-molecule dubbele strengen heeft die zich om elkaar heen wentelen en een dubbele spiraalstructuur vormen. In het algemeen bestaat het genetisch materiaal in de meeste natuurlijke organismen uit dubbele strengen van spiraalvormige DNA’s, waarbij sommige strengen uit enkele DNA-strengen bestaan en andere uit enkele of dubbele strengen RNA’s. DNA-componenten of nucleotiden bestaan uit stikstofhoudende basen, fosfaatgroepen en desoxyribosegroepen. De twee letters zijn gestructureerd als de ruggengraat van elke DNA-molecule, waarbij elk basenpaar van elke streng door een waterstofbrug met elkaar wordt verbonden. DNA-nucleotiden bestaan uit vier soorten basen, waaronder adenine (A), cytosine (C), guanine (G) en thymine (T) (figuur 2), terwijl ribonucleïnezuur (RNA) vier soorten basen heeft, waaronder adenine (A), cytosine (C), guanine (G), en uracil (U) in plaats van thymine (T). Adenine (A) en guanine (G) zijn purines, terwijl cytosine (C), thymine (T) en uracil (U) pyrimidines zijn. In DNA molecules, the base-pairing rule is that A pairs with T, and G pairs with C (Figures 3 and 4) .
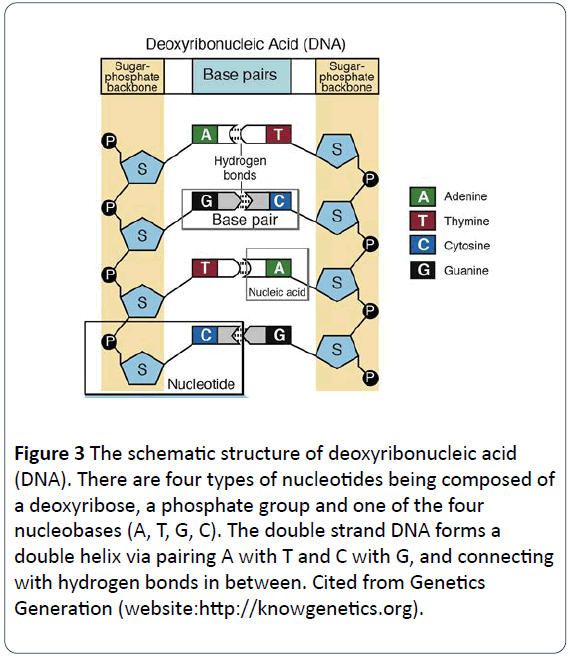
Figure 3: The schematic structure of deoxyribonucleic acid (DNA). There are four types of nucleotides being composed of a deoxyribose, a phosphate group and one of the four nucleobases (A, T, G, C). The double strand DNA forms a double helix via pairing A with T and C with G, and connecting with hydrogen bonds in between. Cited from Genetics Generation (website:https://knowgenetics.org).
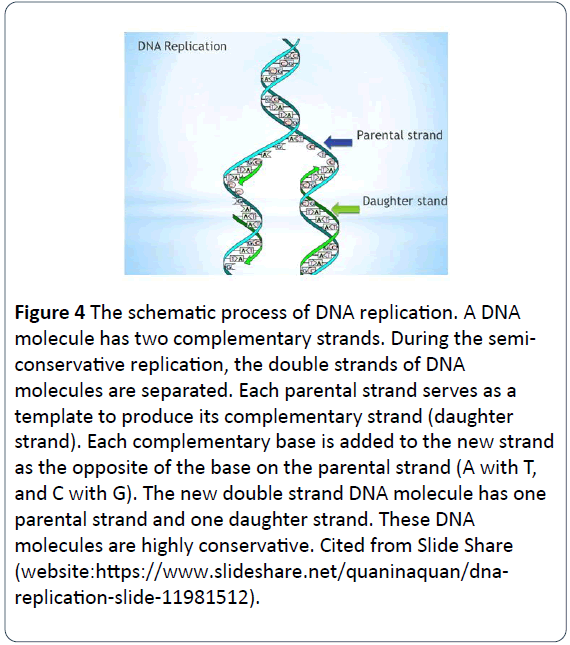
Figure 4: Het schematische proces van DNA replicatie. Een DNA-molecuul heeft twee complementaire strengen. Tijdens de semiconservatieve replicatie worden de dubbele strengen van DNA-moleculen gescheiden. Elke ouderlijke streng dient als sjabloon om zijn complementaire streng (dochterstreng) te produceren. Elke complementaire base wordt aan de nieuwe streng toegevoegd als het tegenovergestelde van de base op de ouderlijke streng (A met T, en C met G). Het nieuwe dubbelstrengs DNA-molecuul heeft één ouderstreng en één dochterstreng. Deze DNA-moleculen zijn zeer conservatief. Aangehaald van Slide Share (website:https://www.slideshare.net/quaninaquan/dnareplication- slide-11981512).
Het proces is dat een dubbelstrengs DNA-molecuul afwikkelt waarbij elk van de twee ouderstrengen wordt gescheiden en als een ouderlijk sjabloon voor de synthese van nieuwe dochter-DNA-moleculen fungeert. De complementaire nucleotiden worden aan de dochterstreng toegevoegd, met fosfaten en desoxyribosen om de ruggengraat van de nieuwe nucleotiden te vormen en nieuwe basen om te paren aan het tegenovergestelde van de basen op de ouderstreng via de basenpaarregel (A paart aan T, en G paart aan C) en om op hun plaats te houden met waterstofbruggen . Uiteindelijk heeft elk van de nieuwe dubbelstrengs DNA-moleculen één ouderstreng en één dochterstreng. De DNA-moleculen repliceren in dit semi-conservatieve model, waardoor genetisch DNA behouden en constant blijft, en van de ene generatie op de andere overgaat (figuur 5) .
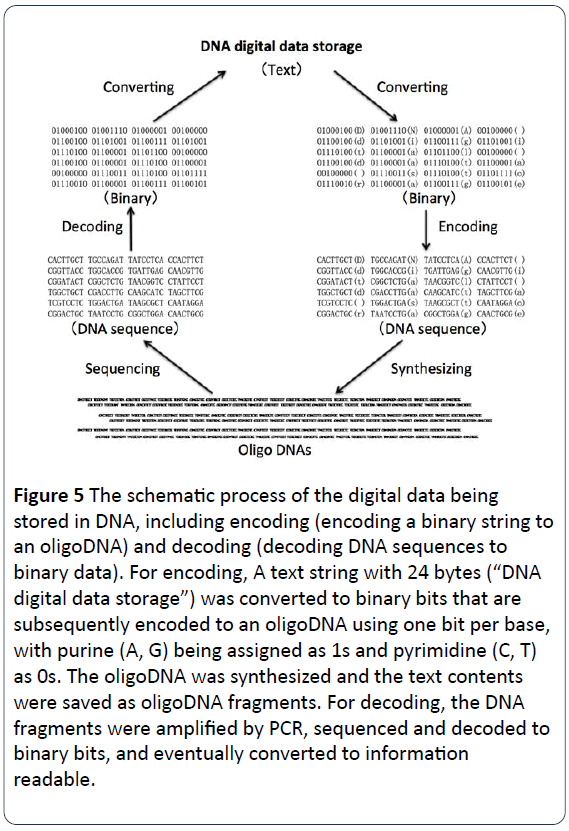
Figuur 5: Het schematische proces van de digitale gegevens die in DNA worden opgeslagen, inclusief coderen (coderen van een binaire tekenreeks naar een oligoDNA) en decoderen (decoderen van DNA-sequenties naar binaire gegevens). Voor het coderen werd een tekststring van 24 bytes (“opslag van digitale DNA-gegevens”) omgezet in binaire bits die vervolgens werden gecodeerd in een oligoDNA met één bit per base, waarbij purine (A, G) als 1s en pyrimidine (C, T) als 0s werden toegewezen. Het oligoDNA werd gesynthetiseerd en de tekstinhoud werd opgeslagen als oligoDNA-fragmenten. Voor het decoderen werden de DNA-fragmenten geamplificeerd door PCR, gesequenced en gedecodeerd tot binaire bits, en uiteindelijk omgezet in leesbare informatie.
Het proces voor de opslag van digitale DNA-gegevens
Het proces voor de opslag van digitale DNA-gegevens is het coderen en decoderen van binaire gegevens naar en van gesynthetiseerde DNA-strengen. De teksten, getallen, afbeeldingen en andere leesbare of zichtbare gegevens worden eerst omgezet in binaire talen met 0 en 1, en vervolgens gecodeerd naar DNA-nucleotidesequenties, met de vier basen (A, C, G, T) in plaats van 0 en 1 . Bijvoorbeeld, een hoofdletter “D” is “01000100” in binair, een kleine letter “d” is “01100100” met een spatie ” ” is “00100000” (figuur 1). In de figuren 3 en 5 werd de zin “opslag van digitale DNA-gegevens” omgezet in een binaire versie om binaire codes met 24 bytes te verkrijgen. Vervolgens worden binaire codes (binaire bits) gecodeerd in DNA-codes. Elk van de vier basen (A, C, G en T) moet worden toegewezen als 1 of 0. Bijvoorbeeld, purine (A, G) wordt toegewezen als 1s, terwijl pyrimidine (C, T) als 0s wordt toegewezen. Of, de twee basen G en T worden toegewezen als 1s, terwijl de andere twee A en C 0s zijn. Zoals blijkt uit figuur 4 is voor het coderen van de gegevens een tekststring van 24 bytes (“digitale DNA-gegevensopslag”) omgezet in binaire bits die vervolgens zijn gecodeerd in een oligo-DNA met gebruikmaking van 1 bit per base, waarbij purine (A, G) worden toegewezen als 1s en pyrimidine (C, T) als 0s. Het oligo-DNA werd chemisch gesynthetiseerd en de tekstinhoud werd vervolgens opgeslagen als oligo-DNA-fragmenten voor opslag op lange termijn. Voor het decoderen van de gegevens werden de DNA-fragmenten geamplificeerd door PCR, gesequenced en gedecodeerd naar binaire bits, één keer per dag, moeten de gegevens worden opgehaald om binaire gegevens leesbaar te maken. Uiteindelijk, het lezen van de gegevens van DNA-sequentie bibliotheek is de sequentie van de unieke DNA-moleculen, het omzetten van de sequentie-informatie in de oorspronkelijke digitale gegevens als nodig of vereiste (figuur 5) .
De voordelen van DNA-gegevens opslagmedium
Zoals hierboven vermeld, zijn de wereldwijde gegevens sterk toegenomen in het exponentiële tempo. De traditionele media kunnen niet voldoende omgaan met de eis van de grote gegevensopslag. DNA kan dienen als een mogelijk medium voor de opslag van digitale gegevens, met zijn potentiële voordelen zoals hoge dichtheid, hoge replicatie-efficiëntie, duurzaamheid en stabiliteit op lange termijn (https:// www.scientificworldinfo.com) . DNA kan op zijn theoretische maximumcapaciteit ongeveer twee bits per nucleotide coderen . Een volledig datacentrum dat in 2011 door IBM is gebouwd, heeft ongeveer 100 petabytes (PB’s) aan gegevensopslagcapaciteit. Dankzij de hoge dichtheid kan DNA als medium voor gegevensopslag een grote hoeveelheid gegevens opslaan op een klein oppervlak. Een enkele gram DNA kan op zijn theoretisch maximum ongeveer 200 PBs aan gegevens opslaan, bijna twee keer zoveel als het hele IBM-datacenter. Met andere woorden, alle informatie die over de hele wereld is opgenomen, kan worden opgeslagen in enkele kilo’s DNA, of gelijk aan slechts één schoenendoos vergeleken met de behoefte aan miljoenen grote gegevensopslagcentra voor traditionele media .
Data-gecodeerd DNA-medium is in staat tot opslag op lange termijn dankzij de hoge duurzaamheid . DNA kan duizenden jaren in een koude, droge en donkere omgeving worden bewaard. Zelfs onder slechtere omstandigheden is de halfwaardetijd van DNA tot wel honderd jaar. DNA kan stabiel blijven bij lage en hoge temperaturen, met een breed bereik van -800°C tot 800°C. DNA-media kunnen gegevens ook beter beveiligen dan traditionele digitale gegevensdragers. Hoewel nieuwe gegevens exponentieel toenemen, worden de meeste opgeslagen in archieven voor langdurige opslag. Deze koude gegevens zullen niet onmiddellijk worden teruggehaald of vaak worden gebruikt. Ze opslaan in DNA-media is dus eenvoudig, handig en kostenloos. Een ander voordeel is dat DNA in hoge mate geconserveerd is. De natuurlijke DNA’s kunnen zichzelf nauwkeurig repliceren met een hoge efficiëntie en altijd volgens de basenpaarregel (A met T, C met G) (figuur 3) . Het DNA-medium kan dus gedurende lange tijd een hoge betrouwbaarheid van de gegevens bewaren.
De uitdagingen voor DNA-gegevensopslagmedium
Gebaseerd op zijn unieke kenmerken en vergeleken met de traditionele media, zou DNA een potentieel en veelbelovend medium kunnen zijn voor digitale gegevensopslag. Er is echter nog een lange weg te gaan voordat DNA commercieel kan worden toegepast. Er zijn verschillende uitdagingen, zoals de hoge kosten, de lage verwerkingscapaciteit, de beperkte toegang tot gegevensopslag, de korte synthetische oligo DNA-fragmenten, het foutenpercentage bij de synthese en de sequencing.
Het gebruik van DNA voor gegevensopslag is veel duurder dan de andere traditionele media zoals tape, schijf en HDD (harde schijf) (https://www.scientificworldinfo.com) . Momenteel kost het coderen en decoderen van gegevens bijna 15.000 dollar per megabyte (MB). Ondertussen is de huidige technologie in DNA-synthese beperkt, met slechts korte oligo DNA-sequenties die kunnen worden gesynthetiseerd. De maximale lengte van elk oligo DNA-fragment is beperkt tot enkele honderden nucleotiden. Aldus, om één enkel gearchiveerd bestand op te slaan, kunnen in het bijzonder 1 groot bestand honderdduizenden oligo DNA’s nodig hebben. En ook is het tijdrovend om gegevens in te schrijven in en op te halen uit oligo DNA’s, met de betrokkenheid van meerdere stappen waaronder het omzetten van gegevens naar binair, het coderen van binair naar oligo DNA, het synthetiseren en opslaan van DNA-sequenties, en het ophalen van unieke sequenties uit de DNA-opslagbibliotheek, het sequencen en decoderen, en uiteindelijk het omzetten van binair naar gegevens die leesbaar zijn. De traditionele media zoals schijf en tape hebben hun logische adresseringsinformatie, maar oligo DNA’s hebben dat niet. Het is dus zeer moeilijk om de unieke gecodeerde DNA-sequentie die wij verwachten te hebben, te adresseren. Ondertussen is willekeurige toegang tot op DNA gebaseerde gegevensopslag belangrijk, maar oligo DNA’s hebben geen mogelijkheid tot willekeurige toegang. Via de huidige benaderingen is alleen bulktoegang beschikbaar voor DNA-gegevensopslag. De volledige op DNA gebaseerde gegevensopslag moet worden gesorteerd, gesequenced en gedecodeerd, ook al hoeven we maar één byte te lezen . Daarom moet de juiste primer worden gebruikt om selectief de juiste DNA-sequentie op te halen. Dit zal ook zorgen voor een willekeurige toegang tijdens het sequencen van DNA en het ophalen van gegevens. De sequencing met de unieke primer kan selectief alleen het vereiste oligoDNA lezen, in plaats van de gehele DNA-bibliotheek . En momenteel zijn DNA-synthese en -sequencing niet volledig perfect. Tijdens de DNA-synthese en de sequencing kunnen zich inserts-, deletie-, substitutie- en andere fouten voordoen, met een foutenmarge van ongeveer 1% per nucleotide. De technologie en de kosten van DNA-synthese en sequencing zijn niet geschikt voor de huidige gegevensopslag.
Vanwege de exponentiële toename van wereldwijde gegevens, het gebrek aan voldoende opslagruimte en de behoefte aan innovatieve opslagbenaderingen, wordt DNA als een potentieel gloednieuw medium een hot topic op het gebied van big data-opslag. Met zijn hoge dichtheid, hoge replicatie-efficiëntie, duurzaamheid op lange termijn en stabiliteit, biedt DNA zijn eigen voordelen ten opzichte van de traditionele media voor gegevensopslag. Ondertussen zijn de toepassingen van digitale DNA-gegevensopslag beperkt vanwege de hoge kosten, het gebrek aan willekeurig toegangsmogelijkheid en het tijdrovende coderen en decoderen van gegevens. Gelukkig gaat de vooruitgang op het gebied van DNA-technologie snel vooruit. Om bijvoorbeeld de sequentiebepaling van het eerste menselijke genoom te voltooien, hebben de wetenschappers wereldwijd ongeveer 10-20 jaar samengewerkt, met een totale kostprijs van 3 miljard dollar in 2013 (Human Genome Project (HGP) website: https://www.genome.gov/human-genomeproject).
Conclusie
Nu hebben wetenschappers slechts enkele duizenden dollars en een paar weken nodig om de sequentie van één volledig menselijk genoom te voltooien. En de verwachting is dat het sequencen van één menselijk genoom in de nabije toekomst slechts honderd dollar of minder zal kosten gedurende enkele uren. Er kan dus worden verwacht dat de kosten betaalbaar zullen zijn. Wetenschappers hebben de uitdaging van de willekeurige toegang tot en adressering van informatie opgelost door unieke primers te ontwerpen om selectief de vereiste informatie te adresseren en op te vragen. Om het optreden van fouten te voorkomen, worden de foutcorrigerende metagegevens gecodeerd in oligo DNA-fragmenten. Intussen zijn de single molecule DNA-sequencers uitgevonden en momenteel beschikbaar. Zij zijn handzaam en draagbaar. Zij kunnen de kosten van DNA-sequencing verder drukken en het terugvinden van DNA-informatie vereenvoudigen. Dus, na de vooruitgang in de technologieën van DNA-gegevensopslag, zal DNA dat dient als een medium voor gegevensopslag een gouden kans zijn in dit tijdperk van big data.
- Mayer C, McInroy GR, Murat P, Delft PV, Balasubramanian S (2016) Een epigenetica-geïnspireerd DNA-gebaseerd gegevensopslagsysteem. Angew Chem Int Ed Engl 55: 11144-11148.
- Swati A, Mathuria, F, Bhavani, S, Malathy E, Mahadevan R (2017) A review on various encoding schemes used in digital DNA data storage. Int J Civil Eng Technol 8: 7-10.
- Appuswamy RLK, Barbry P, Antonini M, Madderson O, Freemont P (2019) Archief: Het gebruik van DNA in de DBMS-opslaghiërarchie. CIDR 2019, Biennal Conference on Innovative Data Systems Research, California, USA.
- De Silva PY, GU Ganegoda (2016) Nieuwe trends van digitale dataopslag in DNA. Biomed Res Int pp: 8072463-8072472.
- Panda DM, Baig KA, Swain MJ, Behera A, Dash D (2018) DNA als digitaal informatieopslagapparaat: hoop of hype? Biotech 8: 9-15.
- Chen K, Kong J, Zhu J, Ermann N, Predki P, et al. (2019) Digitale dataopslag met behulp van DNA nanostructuren en solid-state Nanopores. Nano Lett 19: 1210-1215.
- Yazdi S, Gabrys R, Milenkovic O (2017) Draagbare en foutloze dataopslag op basis van DNA. Sci Rep 7: 5011-5013.
- Church GM, Gao Y, Kosuri S (2012) Next-generation digital information storage in DNA. Science 337: 1628-1630.
- Kuang SY, Zhu G, Wang ZL (2018) Triboelectrification-Enabled Self-Powered Data Storage. Adv Sci (Weinh) 5: 1700658.
- Block FE (1987) Analoge en digitale computertheorie. Int J Clin Monit Comput 4: 47-51.
- O’ Driscoll A, Sleator RD (2013) Synthetisch DNA: de volgende generatie van big data opslag. Bioengineered 4: 123-1235.
- Portin P (2014) De geboorte en ontwikkeling van de DNA-theorie van overerving: zestig jaar sinds de ontdekking van de structuur van DNA. J Genet 93: 293-302.
- Leu K, Obermayer B, Rajamani S, Gerland U, Chen IA (2011) The prebiotic evolutionary advantage of transfering genetic information from RNA to DNA. Nucleic Acids Res 39: 8135-8147.
- Burgers PMJ, Kunkel TA (2017) Eukaryotic DNA replication Fork. Annu Rev Biochem 86: 417-438.
- Akram F, Haq I, Ali H, Laghari AT (2018) Trends om digitale data op te slaan in DNA: een overzicht. Mol Biol Rep 45: 1479-1490.
- Organick L, Ang SD, Chen YJ, Lopez R, Yekhanin S, et al. (2018) Random access in large-scale DNA data storage. Nat Biotechnol 36: 242-248.
- Bornholt J, Lopez R, Carmean DM, Ceze L, Seelig G, et al. (2016) A DNA-based archival storage system. ASPLOS 201 (21st ACM International Conference on Architectural Support for Programming Languages and Operating Systems, Atlanta, GA).
- Newman S, Stephenson AP, Willsey M, Nguyen BH, Takahashi CN, et al. (2019) High density DNA data storage library via dehydration with digital microfluidic retrieval. Nat Commun 10: 1706-1710.
- Yazdi SM, Yuan Y, Ma J, Zhao H, Milenkovic O (2015) A rewritable, random-access DNA-based storage system. Sci Rep 5: 14138-14140.
- Ahn T, Ban H, Park H (2018) Het opslaan van digitale informatie in het lang gelezen DNA. Genomics Inform 16: e30-35.
- Bayley H (2017) Single-molecule DNA sequencing: Getting to the bottom of the well. Nat Nanotechnol 12: 1116-1117.