Health Science Journal
Parole chiave
L’archiviazione digitale dei dati; Acido desossiribonucleico (DNA); Binario; Codifica; Decodifica; Sequenziamento
Introduzione
Oggi, è un’era esplosiva di grandi dati. Questi big data esistono e coprono quasi ovunque, dai negozi di alimentari alle banche, dall’offline all’online, dall’accademia all’industria, dall’ospedale alla comunità, dall’organizzazione al governo. L’archiviazione e la gestione dei grandi dati sta diventando una seria preoccupazione. Attualmente, la maggior parte dei dati in tutto il mondo è principalmente memorizzata su supporti magnetici e ottici come HDD (hard disk drive), DISK, CD, nastri, DVD, hard disk portatili e unità streak USB. Tuttavia, la velocità di crescita di questi dati di archiviazione aumenta in modo esplosivo ad un tasso esponenziale. Questi media tradizionali e la loro limitata capacità di archiviazione dei dati non possono soddisfare il requisito del rapido aumento dei dati digitali. Nel frattempo, la durata di conservazione dei dati di questi media è una sfida importante. La loro durata è molto limitata. Questi media durano solo per un tempo molto limitato. Per esempio, i dischi possono durare per diversi anni e i nastri per diversi decenni. Altre memorie elettroniche possono essere mantenute in buone condizioni per diversi decenni. La capacità di memorizzazione dei dati è un altro problema per la memorizzazione di grandi dati digitali. Un CD può contenere diverse centinaia di megabyte (MB) di dati. Un grande disco rigido può memorizzare un paio di terabyte (TB) di dati. Tuttavia, la loro capacità è lontana dal requisito dei dati informativi esplosivi.
Come ha detto Patrizio, ci sono totalmente 33 Zettabytes (ZBs) di dati in tutto il mondo nel 2018 (https://www.networkworld.com), pari a 22 mila miliardi di gigabyte (GBs). Pertanto, una nuova tecnologia di archiviazione e un sistema innovativo sono necessari per soddisfare il requisito di questa era moderna. L’acido desossiribonucleico (DNA), grazie ai suoi vantaggi unici, dovrebbe essere un mezzo ideale per la memorizzazione dei dati digitali. Memorizzare i dati digitali nel DNA non è una storia nuova. In realtà, è stato descritto dal fisico sovietico Mikhail Neiman nel 1960 (https:// www.geneticsdigest.com). Tuttavia, è stata la prima volta a dimostrare che il DNA può memorizzare dati digitali nel 1988. Qui, in primo luogo introduciamo le applicazioni del DNA come un nuovo mezzo di memorizzazione dei dati digitali e poi discuteremo più dettagli in questo campo del DNA che serve come mezzo di memorizzazione dei dati.
Rassegna degli studi precedenti
Il sistema numerico binario
I computer e altri dispositivi elettronici digitali memorizzano i dati e operano con il sistema numerico binario che utilizza solo due numeri digitali o 0 e 1 . I testi sono convertiti in versione binaria nel sistema informatico. A loro volta, i computer operano e calcolano in binario e alla fine convertono le informazioni in testi leggibili. Un byte contiene otto bit costituiti da 0 o 1 e con 28 (256) valori possibili (da 0 a 255), e memorizza una singola lettera (Figura 1 e Tabella 1). Come mostrato nella tabella di conversione ASCII (Tabella 1). Le ventisei lettere con le maiuscole e le minuscole sono convertite tra Lettera, Binario ed Esadecimale. Per memorizzare un file di grandi dimensioni o documento bisogno di dati di memoria molto di più. Una canzone normale può avere bisogno di decine di megabyte, con un paio di gigabyte per memorizzare un film e diversi terabyte per i libri memorizzati in una grande biblioteca. Come mostrato nella tabella 2 sono le dimensioni di misura e di memoria per l’uso del sistema binario dalla più piccola unità “byte” alle grandi unità, tra cui byte (B), kilobyte (KB), megabyte (MB), gigabyte (GB), terabyte (TB), pegabyte (PB), Exabyte (EB), zettabyte (ZB), yottabyte (YB), brontobyte (BB), Geopbyte (GPB) e così via (https://www.geeksforgeeks.org&https://whatsabyte.com). Le unità come brontobyte (BB), Geopbyte (GPB) sono valori enormi inimmaginabili che potrebbero non essere mai usati nel nostro mondo reale (Tabella 2).

Figura 1: La stringa di testo “DNA digital data storage” è stata convertita come bit binari.
Table 1: The conversion ASCII table of the twenty-six letters with the upper and lower cases among letter, binary and hexadecimal.
| Letter | Binary | Hexadecimal | Letter | Binary | Hexadecimal |
|---|---|---|---|---|---|
| A | 1000001 | 41 | a | 1100001 | 61 |
| B | 1000010 | 42 | b | 1100010 | 62 |
| C | 1000011 | 43 | c | 1100011 | 63 |
| D | 1000100 | 44 | d | 1100100 | 64 |
| E | 1000101 | 45 | e | 1100101 | 65 |
| F | 1000110 | 46 | f | 1100110 | 66 |
| G | 1000111 | 47 | g | 1100111 | 67 |
| H | 1001000 | 48 | h | 1101000 | 68 |
| I | 1001001 | 49 | i | 1101001 | 69 |
| J | 1001010 | 4A | j | 1101010 | 6A |
| K | 1001011 | 4B | k | 1101011 | 6B |
| L | 1001100 | 4C | l | 1101100 | 6C |
| M | 1001101 | 4D | m | 1101101 | 6D |
| N | 1001110 | 4E | n | 1101110 | 6E |
| O | 1001111 | 4F | o | 1101111 | 6F |
| P | 1010000 | 50 | p | 1110000 | 70 |
| Q | 1010001 | 51 | q | 1110001 | 71 |
| R | 1010010 | 52 | r | 1110010 | 72 |
| S | 1010011 | 53 | s | 1110011 | 73 |
| T | 1010100 | 54 | t | 1110100 | 74 |
| U | 1010101 | 55 | u | 1110101 | 75 |
| V | 1010110 | 56 | v | 1110110 | 76 |
| W | 1010111 | 57 | w | 1110111 | 77 |
| X | 1011000 | 58 | x | 1111000 | 78 |
| Y | 1011001 | 59 | y | 1111001 | 79 |
| Z | 1011010 | 5A | z | 1111010 | 7A |
*Note: ACSII (American Standard Code for Information Interchange): serial digital codes to represent number, letters, numerals, and other symbols and to be used as a standard format in the computer system.
Table 2: The sizes of measurement and memory.
| Sizes | Byte Magnitude | Units | Storage* |
|---|---|---|---|
| 1 B | 100 | Byte | A character “A”, “1”, “$” |
| 10 B | 101 | ||
| 100 B | 102 | ||
| 1 KB | 103 | Kilo byte | The size for graphics of small websites ranges between 5 and 100 KB |
| 10 KB | 104 | ||
| 100 KB | 105 | ||
| 1 MB | 106 | Mega byte ( 1 MB: 1 million) |
The size for a high resolution JPEG image is about 1-5 MB |
| 10 MB | 107 | The size for a 3-minute song is about 30 MB | |
| 100 MB | 108 | ||
| 1 GB | 109 | Giga byte | The size for a standard DVD drive is about 5 GB |
| 10 GB | 1010 | (1 GB: 1 billion) | |
| 100 GB | 1011 | ||
| 1 TB | 1012 | Tera byte (1 TB: 1 trillion) |
The size for a typical internal HDD is about 2 TB |
| 10 TB | 1013 | ||
| 100 TB | 1014 | ||
| 1 PB | 1015 | Peta byte (1 PB: 1 quadrillion) |
Google store over 100 PB of all data in their drivers. |
| 10 PB | 1016 | ||
| 100 PB | 1017 | ||
| 1 EB | 1018 | Exa byte (1 EB: 1 quintillion) |
Several hundred EBs of data are transferred over global internet per year Facebook built an entire data center to store 1 EB of data in 2013 |
| 10 EB | 1019 | ||
| 100 EB | 1020 | ||
| 1 ZB | 1021 | Zetta byte (1 ZB: 1 sextillion) |
33 ZBs of global data in 2018. 160-180 ZBs of data is predicted in 2025. |
| 10 ZB | 1022 | ||
| 100 ZB | 1023 | ||
| 1 YB | 1024 | Yotta byte (1 YB: 1 septillion) |
1YB = 1 million EBs 1 YB = Size of the entire World Wide Web |
| 10 YB | 1025 | ||
| 100 YB | 1026 | ||
| 1 BB | 1027 | Bronto byte (1 BB: 1 octillion) |
1BB equals to 1 million ZBs The only thing there is to say about a Brontobyte is that it is a 1 followed by 27 zeros! |
| 10 BB | 1028 | ||
| 100 BB | 1029 | ||
| 1 GPB | 1030 | Geop byte (1 GPB: 1 nonillion) | 1, No one knows why this term was created. It is highly doubtful that anyone alive today will EVER see a Geopbyte hard drive. |
The digital data storage
Digital Data Storage (DDS) was introduced and developed in 1980s. It is a computer-based data storage technology that is based on the Digital Audio Tape (DAT) format. These digital data were stored on the silicon-based chips. Silicon is the primary material of most semiconductor and microelectronic chips. Il silicio puro per la memoria si trova raramente in natura. Si prevede che tutto il silicio di grado microchip in tutto il mondo si esaurirà nel prossimo futuro. Inoltre, la legge di Moore (il numero di transistor ospitati sui circuiti integrati è quasi raddoppiato ogni due anni, o più transistor i chip girano più velocemente con più transistor) sta arrivando alla fine. Così, i chip non possono ospitare ulteriori transistor e raggiungeranno il limite della loro capacità.
Nel frattempo, la maggior parte degli attuali dati digitali sono memorizzati nei tradizionali supporti magnetici, ottici e altri come HDD (hard disk drive) e CD. Oltre alla loro limitata capacità di memorizzazione dei dati, questi supporti possono essere conservati per un tempo molto limitato. Sono sensibili all’ambiente o alla condizione di conservazione dei dati. Qualsiasi cambiamento ambientale e condizionale come l’esposizione magnetica, l’alta umidità, l’alta temperatura, i danni meccanici, possono eventualmente provocare il danneggiamento di questi media o la loro perdita di dati. E l’uso frequente può anche portare al loro danno o alla perdita di dati. E inoltre, per immagazzinare la grande quantità di dati e per soddisfare il requisito dell’aumento esplosivo dei dati, abbiamo bisogno di una grande quantità di tali media come dischi, CD, DVP, hard disk. Questi porteranno a costi elevati e richiederanno molto tempo.
Simultaneamente, l’aumento dei dati digitali e il requisito dell’archiviazione dei dati stanno crescendo ad un tasso esponenziale. IBM ha costruito un grande centro con una capacità di immagazzinamento dati di 120 PB nel 2011. Facebook ha costruito un altro centro più grande con la capacità di memorizzare 1 EB (1000 PB) di dati nel 2013. Tutti gli utenti digitali del mondo hanno prodotto oltre 44 exabyte (EB) (44000 PB) di dati al giorno. C’era totalmente 1 zettabyte (ZB, 1000EBs o 1 milione di PBs) di dati prodotti globalmente nel 2010 e 33 zettabytes (ZB) di dati nel 2018, con 150-200 zettabytes (ZBs) previsti nel 2025 (citato dal sito Datanami: https:// www.datanami.com e dal sito Network world: https:// www.networkworld.com). Per immagazzinare questi dati ci vorrebbero centinaia di migliaia di enormi centri spaziali. Nel 2018, Facebook ha avuto un totale di 15 sedi di data center nel 2018, con altri nuovi centri annunciati. Costruiranno altri quattro data center in Nebraska, composti da sei grandi edifici con spazio di datastoring oltre 2,6 milioni di piedi quadrati. In ogni caso, lo spazio di archiviazione dei dati non potrà mai catturare l’aumento esponenziale dei dati. Anche gli attuali supporti di memorizzazione non possono soddisfare il requisito di memorizzazione. C’è un bisogno urgente di sviluppare una tecnologia di immagazzinamento dati di nuova generazione invece dell’attuale immagazzinamento dati basato sul silicio. Con le sue caratteristiche uniche e i suoi vantaggi potenziali, l’acido desossiribonucleico (DNA) come possibile supporto di archiviazione digitale dei dati sta entrando nella fase centrale dell’archiviazione dei dati.
Le informazioni di base dell’acido desossiribonucleico (DNA)
Nel 1953, i dottori Crick e Watson hanno rivelato che una molecola di DNA ha doppi filamenti che si avvolgono l’uno intorno all’altro e formano una struttura a doppia elica. In generale, il materiale genetico nella maggior parte degli organismi naturali è costituito da doppi filamenti di DNA elicoidali, mentre alcuni sono a singolo filamento di DNA e altri sono a singolo o doppio filamento di RNA. I componenti del DNA o nucleotidi sono costituiti da basi azotate, gruppi fosfato e gruppi desossiribosio. Le due lettere sono strutturate come la spina dorsale di ogni molecola di DNA, con ogni coppia di basi di ogni filamento da collegare con un legame idrogeno. I nucleotidi del DNA sono composti da quattro tipi di basi, tra cui adenina (A), citosina (C), guanina (G) e timina (T) (Figura 2), mentre l’acido ribonucleico (RNA) ha quattro tipi di basi, tra cui adenina (A), citosina (C), guanina (G) e uracile (U) invece di timina (T). Adenina (A) e guanina (G) sono purine, mentre citosina (C), timina (T) e uracile (U) sono pirimidine. In DNA molecules, the base-pairing rule is that A pairs with T, and G pairs with C (Figures 3 and 4) .
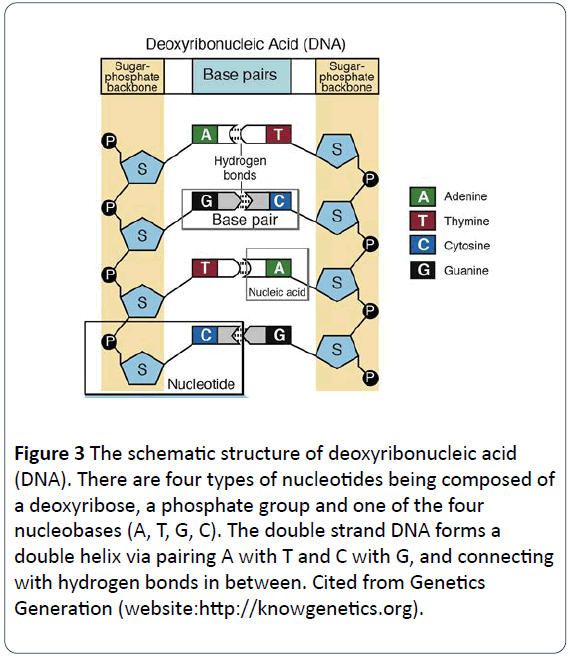
Figure 3: The schematic structure of deoxyribonucleic acid (DNA). There are four types of nucleotides being composed of a deoxyribose, a phosphate group and one of the four nucleobases (A, T, G, C). The double strand DNA forms a double helix via pairing A with T and C with G, and connecting with hydrogen bonds in between. Cited from Genetics Generation (website:https://knowgenetics.org).
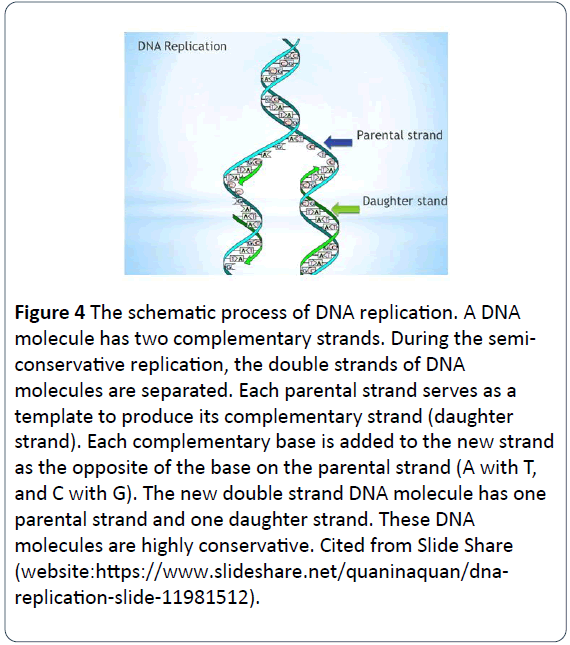
Figure 4: Il processo schematico della replicazione del DNA. Una molecola di DNA ha due filamenti complementari. Durante la replicazione semiconservativa, i doppi filamenti delle molecole di DNA sono separati. Ogni filamento parentale serve da modello per produrre il suo filamento complementare (filamento figlio). Ogni base complementare viene aggiunta al nuovo filamento come l’opposto della base sul filamento parentale (A con T, e C con G). La nuova molecola di DNA a doppio filamento ha un filamento genitore e un filamento figlio. Queste molecole di DNA sono altamente conservative. Citato da Slide Share (sito web:https://www.slideshare.net/quaninaquan/dnareplication- slide-11981512).
Il processo è che una molecola di DNA a doppio filamento si snoda con ciascuno dei due filamenti parentali che vengono separati e agisce come template parentale per la sintesi di nuove molecole di DNA figlie. I nucleotidi complementari vengono aggiunti al filamento figlio, con fosfati e deossiribosi per formare la spina dorsale dei nuovi nucleotidi e nuove basi da accoppiare con l’opposto delle basi sul filamento genitore tramite la regola di accoppiamento delle basi (A che si accoppia con T, e G che si accoppia con C) e da tenere in posizione con legami idrogeno. Alla fine, ciascuna delle nuove molecole di DNA a doppio filamento ha un filamento genitore e un filamento figlio. Le molecole di DNA si replicano in questo modello semi-conservativo, mantengono il DNA genetico conservativo e costante, e passano da una generazione all’altra (Figura 5).
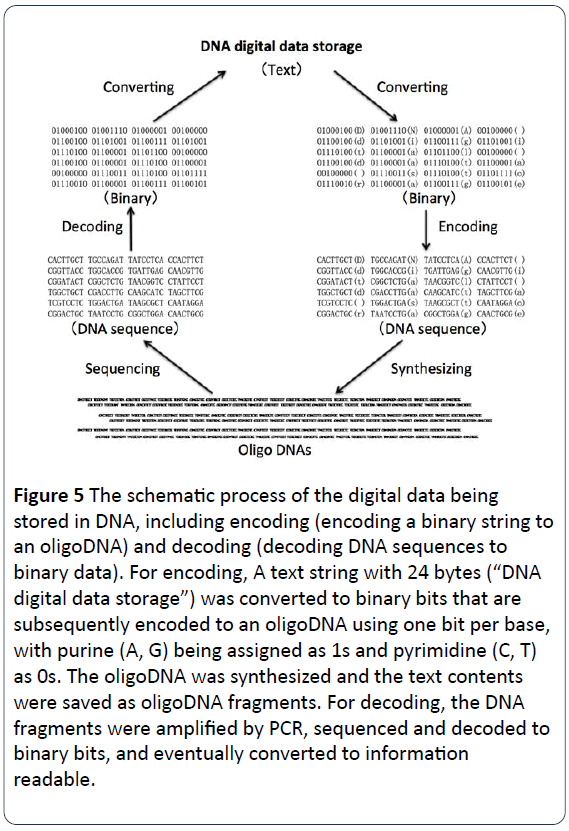
Figura 5: Il processo schematico dei dati digitali memorizzati nel DNA, compresa la codifica (codifica di una stringa binaria in un oligoDNA) e decodifica (decodifica di sequenze di DNA in dati binari). Per la codifica, una stringa di testo con 24 byte (“memorizzazione dei dati digitali nel DNA”) è stata convertita in bit binari che sono successivamente codificati in un oligoDNA utilizzando un bit per base, con le purine (A, G) assegnate come 1s e le pirimidine (C, T) come 0s. L’oligoDNA è stato sintetizzato e il contenuto del testo è stato salvato come frammenti di oligoDNA. Per la decodifica, i frammenti di DNA sono stati amplificati tramite PCR, sequenziati e decodificati in bit binari, e infine convertiti in informazioni leggibili.
Il processo di archiviazione dei dati digitali del DNA
Il processo di archiviazione dei dati digitali del DNA consiste nel codificare e decodificare i dati binari in e da filamenti di DNA sintetizzati. I testi, i numeri, le immagini e altri leggibili o visibili prima vengono convertiti in linguaggi binari con 0 e 1, e poi codificati in sequenze nucleotidiche di DNA, con le quattro basi (A, C, G, T) invece di 0 e 1. Per esempio, una lettera maiuscola “D” è “01000100” in binario, una lettera minuscola “d” è “01100100” con uno spazio vuoto ” ” è “00100000” (Figura 1). Nelle figure 3 e 5, la frase “archiviazione digitale dei dati del DNA” è stata convertita in versione binaria per ottenere codici binari con 24 byte. Poi, i codici binari (bit binari) sono codificati in codici DNA. Ognuna delle quattro basi (A, C, G e T) deve essere assegnata come 1 o 0. Per esempio, la purina (A, G) è assegnata come 1s, mentre la pirimidina (C, T) è come 0s. Oppure, le due basi G e T sono assegnate come 1s, mentre le altre due A e C sono 0s. Come mostrato nella Figura 4, per la codifica dei dati, una stringa di testo con 24 byte (“archiviazione digitale dei dati del DNA”) è stata convertita in bit binari che sono successivamente codificati in un oligo DNA usando un 1 bit per base, con purine (A, G) assegnate come 1s e pirimidine (C, T) come 0s. L’oligo DNA è stato sintetizzato chimicamente e il contenuto del testo è stato poi salvato come frammenti di oligo DNA per la conservazione a lungo termine. Per la decodifica dei dati, i frammenti di DNA sono stati amplificati tramite PCR, sequenziati e decodificati in bit binari una volta al giorno, abbiamo bisogno di recuperare i dati per produrre dati binari da leggere. Alla fine, la lettura dei dati dalla libreria di sequenze di DNA è quella di sequenziare le molecole di DNA uniche, convertire le informazioni di sequenziamento nei dati digitali originali come necessario o requisito (Figura 5).
I vantaggi del supporto di archiviazione dei dati del DNA
Come menzionato sopra, i dati globali sono fortemente aumentati al tasso esponenziale. I media tradizionali non possono affrontare sufficientemente il requisito della conservazione dei dati di grandi dimensioni. Il DNA può servire come un possibile mezzo di archiviazione dei dati digitali, con i suoi potenziali vantaggi come l’alta densità, l’alta efficienza di replicazione, la durata e la stabilità a lungo termine (https:// www.scientificworldinfo.com). Il DNA alla sua capacità massima teorica può codificare circa due bit per nucleotide. Un intero centro dati costruito da IBM nel 2011 ha circa 100 petabyte (PB) di capacità di archiviazione dati. Tuttavia, a causa della sua alta densità, il DNA che agisce come un mezzo di memorizzazione dei dati può memorizzare una grande quantità di dati in una piccola dimensione. Un singolo grammo di DNA al suo massimo teorico può memorizzare circa 200 PB di dati, quasi il doppio di quello dell’intero centro dati IBM. In altre parole, tutte le informazioni registrate in tutto il mondo possono essere memorizzate in diversi chilogrammi di DNA, o pari a una sola scatola da scarpe rispetto al requisito di milioni di grandi centri di archiviazione dati per i media tradizionali .
Il mezzo DNA codificato dai dati è in grado di memorizzare a lungo termine grazie alla sua elevata durata. Il DNA può durare per migliaia di anni in luoghi freddi, asciutti e bui. Anche in ambienti peggiori, l’emivita del DNA è fino a cento anni. Il DNA può rimanere stabile a bassa temperatura o ad alta temperatura, con un’ampia gamma da -800°C a 800°C. I supporti di DNA possono anche proteggere i dati più dei tradizionali supporti di dati digitali. Anche se i nuovi dati stanno aumentando ad un tasso esponenziale, la maggior parte di essi sono salvati in archivi per la conservazione a lungo termine. Questi dati freddi non saranno recuperati immediatamente o utilizzati frequentemente. Quindi, memorizzarli in supporti di DNA è semplice, conveniente e senza costi. Un altro vantaggio è che il DNA è altamente conservato. I DNA naturali possono replicarsi accuratamente ad alta efficienza e sempre con la regola di accoppiamento delle basi (A con T, C con G) (Figura 3). Così, il mezzo del DNA può mantenere la fedeltà dei dati per un lungo periodo di tempo.
Le sfide per il mezzo di archiviazione dei dati del DNA
In base alle sue caratteristiche uniche e rispetto ai media tradizionali, il DNA potrebbe essere il potenziale e promettente mezzo per l’archiviazione dei dati digitali. Tuttavia, c’è ancora molta strada da fare prima che il DNA possa essere applicato commercialmente. Le sfide che dobbiamo affrontare esistono in vari aspetti, tra cui l’alto costo, il basso rendimento, l’accesso limitato all’immagazzinamento dei dati, i brevi frammenti sintetici di oligo DNA, il tasso di errore nella sintesi e nel sequenziamento.
L’uso del DNA nell’immagazzinamento dei dati è molto più costoso degli altri media tradizionali come nastro, disco e HDD (hard disk drive) (https://www.scientificworldinfo.com). Attualmente, codificare e decodificare i dati costa quasi 15.000 dollari per megabyte (MB). Nel frattempo, la tecnologia attuale nella sintesi del DNA è limitata, con solo brevi sequenze di DNA oligo da sintetizzare. La lunghezza massima di ogni frammento di DNA oligo è limitata a diverse centinaia di nucleotidi. Così, per memorizzare un singolo file archiviato, in particolare 1 file di grandi dimensioni può avere bisogno di centinaia di migliaia di oligo DNA. E inoltre, è lungo per i dati da scrivere in e recuperare da oligo DNA, con il coinvolgimento di più passaggi tra cui la conversione dei dati in binario, la codifica binaria in oligo DNA, la sintesi e la memorizzazione di sequenze di DNA, e il recupero di sequenze uniche dalla biblioteca di archiviazione del DNA, il sequenziamento e la decodifica, e infine la conversione binaria in dati leggibili. I media tradizionali come il disco e il nastro hanno le loro informazioni di indirizzamento logico, tuttavia, i DNA oligo non le hanno. Quindi, è molto difficile indirizzare l’unica sequenza di DNA codificata che ci aspettiamo di avere . Nel frattempo, l’accesso casuale all’archiviazione dei dati basata sul DNA è importante, tuttavia, i DNA oligo non hanno capacità di accesso casuale. Attraverso gli approcci attuali, solo l’accesso in massa è disponibile per l’archiviazione dei dati del DNA. L’intera memoria di dati basata sul DNA deve essere ordinata, sequenziata e decodificata dalla memoria di dati del DNA anche se abbiamo solo bisogno di leggere un singolo byte. Pertanto, è necessario il giusto primer utilizzato per recuperare selettivamente la giusta sequenza di DNA. Questo fornirà anche un accesso casuale durante il sequenziamento del DNA e il recupero dei dati. Il sequenziamento con il primer unico può leggere selettivamente solo l’oligoDNA richiesto, piuttosto che l’intera libreria di DNA. E attualmente, la sintesi del DNA e il sequenziamento non sono completamente perfetti. Durante la sintesi del DNA e il sequenziamento, si possono verificare errori di inserimento, cancellazione, sostituzione e altri errori, con un tasso di errore di circa l’1% per nucleotide. La tecnologia e il costo della sintesi e del sequenziamento del DNA non sono adatti all’attuale archiviazione dei dati.
Per quanto riguarda il supporto di archiviazione dei dati del DNA
A causa dell’aumento esponenziale dei dati globali, la mancanza di spazi di archiviazione sufficienti e la necessità di approcci di archiviazione innovativi, il DNA come potenziale nuovo supporto sta diventando un argomento caldo nel campo dell’archiviazione dei grandi dati. Con l’alta densità, l’alta efficienza di replica, la durata a lungo termine e la stabilità, il DNA mostra i propri vantaggi rispetto ai tradizionali mezzi di archiviazione dei dati. Nel frattempo, le applicazioni di archiviazione dei dati digitali del DNA sono state limitate a causa del costo elevato, la mancanza di capacità di accesso casuale, che richiede tempo nella codifica e decodifica dei dati. Fortunatamente, il progresso nel campo della tecnologia del DNA sta avanzando rapidamente. Per esempio, per completare il sequenziamento del primo genoma umano, gli scienziati mondiali hanno collaborato e lavorato insieme per circa 10-20 anni, con un costo totale di 3 miliardi di dollari nel 2013 (sito web Human Genome Project (HGP): https://www.genome.gov/human-genomeproject).
Conclusione
Oggi, gli scienziati hanno solo bisogno di alcune migliaia di dollari e un paio di settimane per finire la sequenza di un intero genoma umano. E ci si aspetta che il sequenziamento di un genoma umano costi solo centinaia di dollari o meno per diverse ore nel prossimo futuro. Quindi, ci si può aspettare che il costo sia accessibile. Per l’accesso casuale e l’indirizzamento delle informazioni, gli scienziati hanno risolto questa sfida progettando i primer unici per indirizzare e recuperare selettivamente le informazioni richieste. Per evitare il verificarsi di errori, i metadati di correzione degli errori sono codificati in frammenti di DNA oligo. Nel frattempo, i sequenziatori di DNA a singola molecola sono stati inventati e sono attualmente disponibili. Sono maneggevoli e portatili. Possono ridurre ulteriormente il costo del sequenziamento del DNA e semplificare il recupero delle informazioni sul DNA. Quindi, seguendo i progressi nelle tecnologie di archiviazione dei dati del DNA, il DNA che serve come mezzo di archiviazione dei dati sarà un’opportunità d’oro in questa era di big data.
- Mayer C, McInroy GR, Murat P, Delft PV, Balasubramanian S (2016) Un sistema di archiviazione dati basato sul DNA ispirato all’epigenetica. Angew Chem Int Ed Engl 55: 11144-11148.
- Swati A, Mathuria, F, Bhavani, S, Malathy E, Mahadevan R (2017) Una revisione su vari schemi di codifica utilizzati nella memorizzazione digitale dei dati del DNA. Int J Civil Eng Technol 8: 7-10.
- Appuswamy RLK, Barbry P, Antonini M, Madderson O, Freemont P (2019) Archivio: Utilizzo del DNA nella gerarchia di archiviazione del DBMS. CIDR 2019, Biennal Conference on Innovative Data Systems Research, California, USA.
- De Silva PY, GU Ganegoda (2016) Nuove tendenze di archiviazione dei dati digitali nel DNA. Biomed Res Int pp: 8072463-8072472.
- Panda DM, Baig KA, Swain MJ, Behera A, Dash D (2018) DNA come dispositivo di archiviazione delle informazioni digitali: speranza o hype? Biotech 8: 9-15.
- Chen K, Kong J, Zhu J, Ermann N, Predki P, et al. (2019) Archiviazione di dati digitali utilizzando nanostrutture di DNA e nanopori a stato solido. Nano Lett 19: 1210-1215.
- Yazdi S, Gabrys R, Milenkovic O (2017) Archiviazione dati portatile e senza errori basata sul DNA. Sci Rep 7: 5011-5013.
- Church GM, Gao Y, Kosuri S (2012) Archiviazione di informazioni digitali di prossima generazione nel DNA. Science 337: 1628-1630.
- Kuang SY, Zhu G, Wang ZL (2018) Triboelectrification-Enabled Self-Powered Data Storage. Adv Sci (Weinh) 5: 1700658.
- Block FE (1987) Teoria dei computer analogici e digitali. Int J Clin Monit Comput 4: 47-51.
- O’ Driscoll A, Sleator RD (2013) Synthetic DNA: the next generation of big data storage. Bioengineered 4: 123-1235.
- Portin P (2014) La nascita e lo sviluppo della teoria del DNA dell’eredità: sessant’anni dalla scoperta della struttura del DNA. J Genet 93: 293-302.
- Leu K, Obermayer B, Rajamani S, Gerland U, Chen IA (2011) Il vantaggio evolutivo prebiotico del trasferimento di informazioni genetiche da RNA a DNA. Nucleic Acids Res 39: 8135-8147.
- Burgers PMJ, Kunkel TA (2017) Eukaryotic DNA replicazione Fork. Annu Rev Biochem 86: 417-438.
- Akram F, Haq I, Ali H, Laghari AT (2018) Tendenze per memorizzare dati digitali nel DNA: una panoramica. Mol Biol Rep 45: 1479-1490.
- Organick L, Ang SD, Chen YJ, Lopez R, Yekhanin S, et al. (2018) L’accesso casuale nella memorizzazione dei dati del DNA su larga scala. Nat Biotechnol 36: 242-248.
- Bornholt J, Lopez R, Carmean DM, Ceze L, Seelig G, et al. (2016) Un sistema di archiviazione basato sul DNA. ASPLOS 201 (21st ACM International Conference on Architectural Support for Programming Languages and Operating Systems, Atlanta, GA).
- Newman S, Stephenson AP, Willsey M, Nguyen BH, Takahashi CN, et al. (2019) Libreria di archiviazione dati ad alta densità del DNA tramite disidratazione con recupero microfluidico digitale. Nat Commun 10: 1706-1710.
- Yazdi SM, Yuan Y, Ma J, Zhao H, Milenkovic O (2015) Un sistema di archiviazione basato sul DNA riscrivibile e ad accesso casuale. Sci Rep 5: 14138-14140.
- Ahn T, Ban H, Park H (2018) Memorizzazione di informazioni digitali nel DNA di lunga lettura. Genomics Inform 16: e30-35.
- Bayley H (2017) Single-molecule DNA sequencing: Arrivare in fondo al pozzo. Nat Nanotechnol 12: 1116-1117.