ヘルスサイエンスジャーナル
キーワード
デジタルデータ保存、デオキシリボ核酸(DNA)、バイナリ、エンコード、デコード、シーケンス
導入
現在、ビッグデータの時代が爆発的に到来しています。 食料品店から銀行まで、オフラインからオンラインまで、大学から産業まで、病院からコミュニティまで、組織から政府まで、ほとんどあらゆるところにこれらのビッグデータが存在し、カバーしています。 このようなビッグデータの保存と管理は深刻な問題になっています。 現在、世界中のほとんどのデータは、HDD(ハードディスクドライブ)、DISK、CD、テープ、DVD、ポータブルハードドライブ、USBメモリなどの磁気・光学メディアに保存されています。 しかし、これらの保存データの増加速度は指数関数的に増加しています。 このようなデジタルデータの急激な増加に対して、従来のメディアとその限られたデータ保存容量では対応しきれない。 一方、これらのメディアのデータ保存の耐久性は、大きな課題の一つである。 その耐久性は非常に限られています。 これらのメディアは、非常に限られた時間しか持ちこたえられないのです。 例えば、ディスクは数年、テープは数十年の寿命があります。 その他の電子ストレージは、数十年間は良好な状態を保つことができます。 また、大容量のデジタルデータを保存するためには、データ保存の容量も問題となる。 CDは数百メガバイト(MB)のデータを保存することができる。 大容量のハードディスクなら、数テラバイト(TB)のデータを保存できるかもしれない。
パトリツィオが言うように、2018年には世界中で33ゼタバイト (ZB) のデータがあり (https://www.networkworld.com) 、これは22兆ギガバイト (GBs) に相当します。 そのため、この現代に求められる新しいストレージ技術や革新的なシステムが必要とされています。 デオキシリボ核酸(DNA)は、そのユニークな利点から、デジタルデータ保存のための理想的な媒体として期待されている。 DNAにデジタルデータを格納することは、今に始まったことではありません。 実は,1960年代にソ連の物理学者Mikhail Neimanによって報告されている(https:// www.geneticsdigest.com)。 しかし、DNAがデジタル・データを格納できることを初めて実証したのは1988年であった。
これまでの研究のレビュー
二進数システム
コンピューターやその他のデジタル電子機器は、0 と 1 の 2 つのデジタル数字だけを使用する二進数システムでデータを保存し動作します。 テキストは、コンピュータ システムで 2 進数に変換されます。 コンピュータは2進数で動作、計算し、最終的に情報を読み取り可能なテキストに変換します。 1バイトは0か1の8ビットで構成され、0から255までの28(256)個の値を持ち、1つの文字を格納する(図1、表1)。 変換ASCII表(表1)に示すとおりです。 大文字と小文字を含む26文字をLetter、Binary、Hexadecimalに変換している。 大きなファイルや文書を保存するには、より多くのメモリデータが必要です。 普通の曲なら数十メガバイト、映画なら数ギガバイト、図書館の本なら数テラバイトが必要である。 表2に示すように、2進法での計測とメモリのサイズは、最小単位の「バイト」から、バイト(B)、キロバイト(KB)、メガバイト(MB)、ギガバイト(GB)など、大きな単位まである。 テラバイト(TB)、ペガバイト(PB)、エクサバイト(EB)、ゼッタバイト(ZB)、ヨッタバイト(YB)、ブロントバイト(BB)、ジオバイト(GPB)など(https://www.geeksforgeeks.org&https://whatsabyte.com).

“DNA digital data storage” という文字列は、2 進ビットで変換されます。
Table 1: The conversion ASCII table of the twenty-six letters with the upper and lower cases among letter, binary and hexadecimal.
| Letter | Binary | Hexadecimal | Letter | Binary | Hexadecimal |
|---|---|---|---|---|---|
| A | 1000001 | 41 | a | 1100001 | 61 |
| B | 1000010 | 42 | b | 1100010 | 62 |
| C | 1000011 | 43 | c | 1100011 | 63 |
| D | 1000100 | 44 | d | 1100100 | 64 |
| E | 1000101 | 45 | e | 1100101 | 65 |
| F | 1000110 | 46 | f | 1100110 | 66 |
| G | 1000111 | 47 | g | 1100111 | 67 |
| H | 1001000 | 48 | h | 1101000 | 68 |
| I | 1001001 | 49 | i | 1101001 | 69 |
| J | 1001010 | 4A | j | 1101010 | 6A |
| K | 1001011 | 4B | k | 1101011 | 6B |
| L | 1001100 | 4C | l | 1101100 | 6C |
| M | 1001101 | 4D | m | 1101101 | 6D |
| N | 1001110 | 4E | n | 1101110 | 6E |
| O | 1001111 | 4F | o | 1101111 | 6F |
| P | 1010000 | 50 | p | 1110000 | 70 |
| Q | 1010001 | 51 | q | 1110001 | 71 |
| R | 1010010 | 52 | r | 1110010 | 72 |
| S | 1010011 | 53 | s | 1110011 | 73 |
| T | 1010100 | 54 | t | 1110100 | 74 |
| U | 1010101 | 55 | u | 1110101 | 75 |
| V | 1010110 | 56 | v | 1110110 | 76 |
| W | 1010111 | 57 | w | 1110111 | 77 |
| X | 1011000 | 58 | x | 1111000 | 78 |
| Y | 1011001 | 59 | y | 1111001 | 79 |
| Z | 1011010 | 5A | z | 1111010 | 7A |
*Note: ACSII (American Standard Code for Information Interchange): serial digital codes to represent number, letters, numerals, and other symbols and to be used as a standard format in the computer system.
Table 2: The sizes of measurement and memory.
| Sizes | Byte Magnitude | Units | Storage* |
|---|---|---|---|
| 1 B | 100 | Byte | A character “A”, “1”, “$” |
| 10 B | 101 | ||
| 100 B | 102 | ||
| 1 KB | 103 | Kilo byte | The size for graphics of small websites ranges between 5 and 100 KB |
| 10 KB | 104 | ||
| 100 KB | 105 | ||
| 1 MB | 106 | Mega byte ( 1 MB: 1 million) |
The size for a high resolution JPEG image is about 1-5 MB |
| 10 MB | 107 | The size for a 3-minute song is about 30 MB | |
| 100 MB | 108 | ||
| 1 GB | 109 | Giga byte | The size for a standard DVD drive is about 5 GB |
| 10 GB | 1010 | (1 GB: 1 billion) | |
| 100 GB | 1011 | ||
| 1 TB | 1012 | Tera byte (1 TB: 1 trillion) |
The size for a typical internal HDD is about 2 TB |
| 10 TB | 1013 | ||
| 100 TB | 1014 | ||
| 1 PB | 1015 | Peta byte (1 PB: 1 quadrillion) |
Google store over 100 PB of all data in their drivers. |
| 10 PB | 1016 | ||
| 100 PB | 1017 | ||
| 1 EB | 1018 | Exa byte (1 EB: 1 quintillion) |
Several hundred EBs of data are transferred over global internet per year Facebook built an entire data center to store 1 EB of data in 2013 |
| 10 EB | 1019 | ||
| 100 EB | 1020 | ||
| 1 ZB | 1021 | Zetta byte (1 ZB: 1 sextillion) |
33 ZBs of global data in 2018. 160-180 ZBs of data is predicted in 2025. |
| 10 ZB | 1022 | ||
| 100 ZB | 1023 | ||
| 1 YB | 1024 | Yotta byte (1 YB: 1 septillion) |
1YB = 1 million EBs 1 YB = Size of the entire World Wide Web |
| 10 YB | 1025 | ||
| 100 YB | 1026 | ||
| 1 BB | 1027 | Bronto byte (1 BB: 1 octillion) |
1BB equals to 1 million ZBs The only thing there is to say about a Brontobyte is that it is a 1 followed by 27 zeros! |
| 10 BB | 1028 | ||
| 100 BB | 1029 | ||
| 1 GPB | 1030 | Geop byte (1 GPB: 1 nonillion) | 1, No one knows why this term was created. It is highly doubtful that anyone alive today will EVER see a Geopbyte hard drive. |
The digital data storage
Digital Data Storage (DDS) was introduced and developed in 1980s. It is a computer-based data storage technology that is based on the Digital Audio Tape (DAT) format. These digital data were stored on the silicon-based chips. Silicon is the primary material of most semiconductor and microelectronic chips. メモリーグレードの純粋なシリコンは、自然界にはほとんど存在しない。 近い将来、世界中のマイクロチップ用シリコンがすべて枯渇すると予想されている。 また、ムーアの法則(集積回路に搭載されるトランジスタの数は2年ごとにほぼ倍増する、つまりトランジスタの数が多いほどチップの動作速度が速くなる)も終わりを迎えつつある。
一方、現在のデジタル データのほとんどは、HDD (ハード ディスク ドライブ) や CD などの従来の磁気メディアや光学メディアなどに保存されています。
一方、現在のデジタル データのほとんどは、HDD (ハード ディスク ドライブ) や CD などの従来の磁気メディアや光学メディアに保存されています。 また、これらのメディアは、環境やデータの保存状態に敏感です。 磁気への暴露、高湿、高温、機械的な損傷などの環境および条件の変化は、これらのメディアの損傷またはデータの損失をもたらす可能性があります。 また、使用頻度が高い場合も、破損やデータ消失につながる可能性があります。 また、大容量データの保存や爆発的なデータ量の増加に対応するためには、ディスク、CD、DVP、ハードディスクなど、大量のメディアが必要になります。
同時に、デジタルデータの増加とデータストレージの要件は指数関数的に増加しています。 IBMは2011年に120ピコ秒のデータ保存容量を持つ大規模センターを建設しました。 フェイスブックは2013年に1EB(1000PBs)のデータを保存する能力を持つ、さらに大きなセンターを建設した。 世界中のすべてのデジタルユーザーが1日に生み出すデータは44エクサバイト(EBs)(44000PBs)以上。 2010年には全世界で1ゼタバイト(ZB、1000EBsまたは100万PBs)、2018年には33ゼタバイト(ZB)、2025年には150~200ゼタバイト(ZBs)のデータが予測されています(Datanamiウェブサイト: https:// www.datanami.com および Network worldウェブサイト: https:// www.networkworld.com から引用)。 これらのデータを保存するためには、何十万もの巨大な宇宙センターが必要になります。 2018年、Facebookは合計15ヶ所のデータセンターを持ち、さらに新しいセンターが発表されています。 彼らはネブラスカ州にさらに4つのデータセンターを建設する予定で、6つの大きな建物からなり、260万平方フィート以上のデータスタリングスペースを持つ。 何はともあれ、データストレージのスペースは、データの指数関数的な増加を決して捕らえることはできない。 また、現在のストレージメディアでは、ストレージの要件を満たすことができない。 現在のシリコンベースのデータストレージに代わる、新世代のデータストレージ技術の開発が急務となっている。
デオキシリボ核酸 (DNA) の基本情報
1953年、クリック博士とワトソン博士は、DNA分子が互いに巻き付き、二重らせん構造を形成していることを発表しました。 一般に、ほとんどの天然生物の遺伝物質は、らせん状のDNAの二本鎖ですが、一部はDNAの一本鎖、一部はRNAの一本鎖または二本鎖となっています。 DNAの構成要素であるヌクレオチドは、窒素塩基、リン酸基、デオキシリボース基で構成されている。 この2文字が各DNA分子の骨格となり、各鎖から一対の塩基が水素結合でつながる構造になっている。 DNAの塩基はアデニン(A)、シトシン(C)、グアニン(G)、チミン(T)の4種類からなり(図2)、リボ核酸(RNA)はアデニン(A)、シトシン(C)、グアニン(G)、チミン(T)の代わりにウラシル(U)という4種類の塩基からなる(図3)。 アデニン(A)とグアニン(G)はプリンで、シトシン(C)、チミン(T)、ウラシル(U)はピリミジンである。 In DNA molecules, the base-pairing rule is that A pairs with T, and G pairs with C (Figures 3 and 4) .
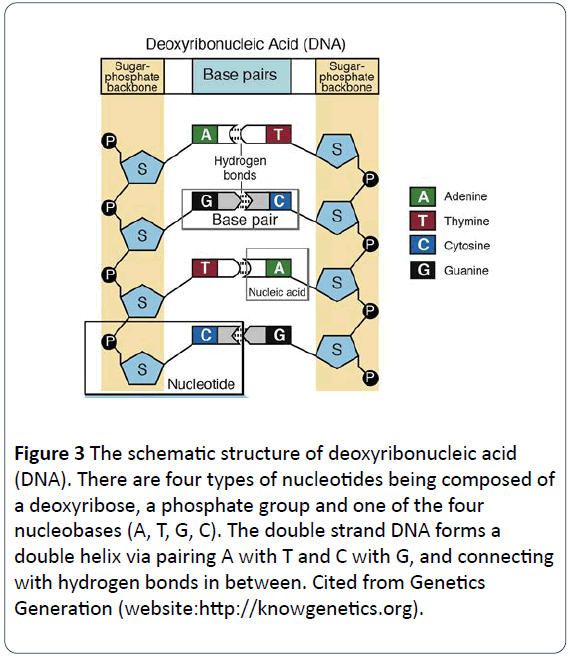
Figure 3: The schematic structure of deoxyribonucleic acid (DNA). There are four types of nucleotides being composed of a deoxyribose, a phosphate group and one of the four nucleobases (A, T, G, C). The double strand DNA forms a double helix via pairing A with T and C with G, and connecting with hydrogen bonds in between. Cited from Genetics Generation (website:https://knowgenetics.org).
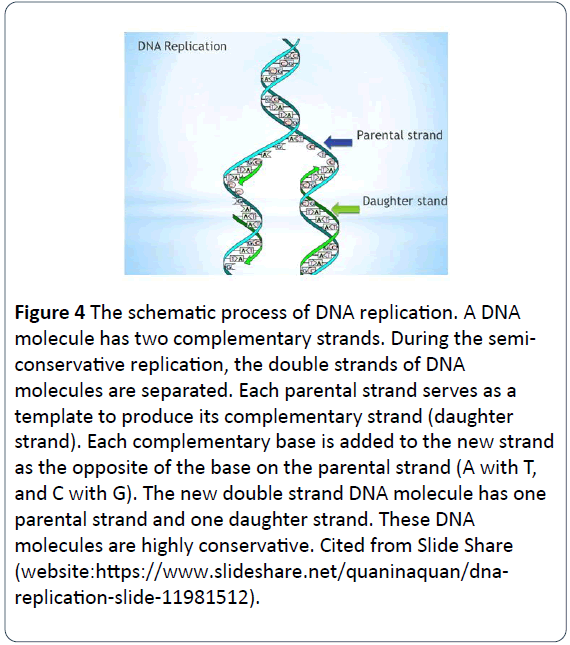
Figure 4: DNA複製の過程を模式的に示したもの。 DNA分子は2本の相補的な鎖を持っている。 半保存的複製の間に、DNA分子の二本鎖は分離される。 それぞれの親鎖は、その相補鎖(娘鎖)を生成するための鋳型となる。 それぞれの相補的な塩基は、親鎖上の塩基の反対側として新しい鎖に付加される(AはTに、CはGに)。 新しい二本鎖DNA分子は、1本の親鎖と1本の娘鎖を持つ。 このようなDNA分子は非常に保存性が高い。 Slide Share (website:https://www.slideshare.net/quaninaquan/dnareplication- slide-11981512) より引用。
プロセスは、2本の親鎖のそれぞれが分離されている状態で二重鎖DNA分子がほどけ、新しい娘DNA分子の合成用の親テンプレートとして機能する、というものです。 娘鎖には、リン酸塩とデオキシリボースが新しいヌクレオチドの骨格を形成し、新しい塩基が塩基対形成規則(AはTと、GはCと対になる)により親鎖の塩基の反対側と対になって、水素結合で固定され、相補的な塩基が追加されます。 最終的に、新しい二本鎖DNA分子は、それぞれ1本の親鎖と1本の娘鎖を持つようになる。 DNA 分子はこの半保存的モデルで複製され、遺伝的 DNA を保守的に一定に保ち、世代から世代へと受け継がれていきます (図 5)。
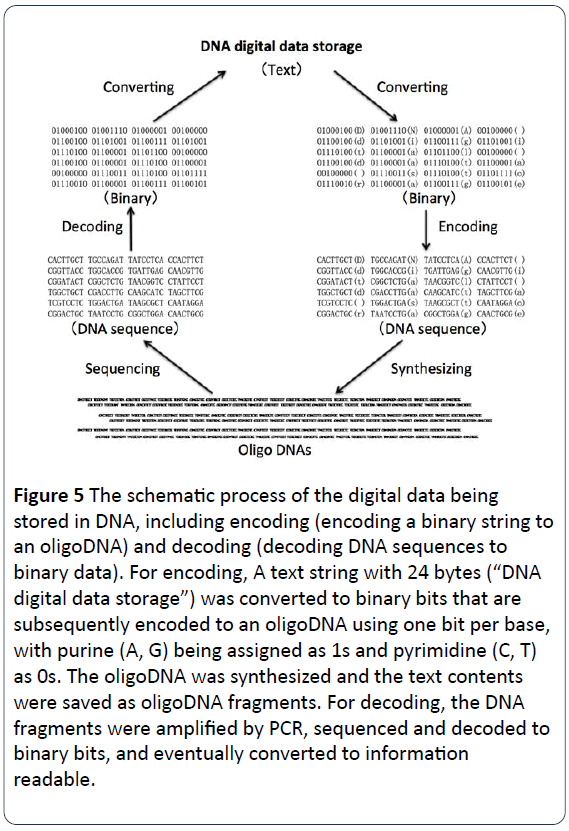
図 5: デジタルデータが DNA に格納される概略プロセス。エンコーディング (2 進文字列からオリゴ DNA にコードする) とデコーディング (DNA 配列からバイナリーデータに復号化) を含む。 エンコードでは、24バイトの文字列(「DNAデジタルデータ記憶装置」)を2値ビットに変換し、その後、プリン(A、G)を1、ピリミジン(C、T)を0として、1塩基あたり1ビットずつ使ってオリゴDNAにエンコードした。 このオリゴDNAを合成し、テキスト内容をオリゴDNAの断片として保存した。 復号化には、DNA断片をPCRで増幅し、塩基配列を決め、2値ビットに復号化し、最終的に情報が読めるように変換した。
DNAデジタルデータの保存プロセス
DNAデジタルデータの保存プロセスは、合成したDNA鎖に2値データを符号化し、復号化することである。 文字や数字、画像など目に見えるものをまず0と1の2進数に変換し、0と1の代わりにA、C、G、Tの4つの塩基を持つDNA塩基配列に符号化します。 例えば、大文字の「D」は2進数で「01000100」、小文字の「d」は「01100100」、空白の「 」は「00100000」である(図1)。 図3、図5では、「DNAデジタルデータ記憶」という文章を2進数に変換し、24バイトの2進数コードを得ています。 そして、バイナリコード(2値ビット)をDNAコードに符号化する。 例えば、プリン(A、G)を1、ピリミジン(C、T)を0として、4つの塩基(A、C、G、T)にそれぞれ1か0を割り当てる。 あるいは、GとTの2塩基を1とし、残りのAとCの2塩基を0として割り当てる。 図4に示すように、24バイトの文字列(「DNAデジタルデータ記憶装置」)を2値ビットに変換し、その後、プリン(A、G)を1s、ピリミジン(C、T)を0sとして、1塩基あたり1bitでオリゴDNAに符号化したものである。 このオリゴDNAを化学的に合成し、テキストコンテンツをオリゴDNA断片として保存し、長期保存を可能にしました。 データ復号化には、DNA断片をPCRで増幅し、塩基配列を決定し、1日1回バイナリビットに復号化し、バイナリデータを読み取れるように出力するためのデータ取り出しが必要である。 最終的に、DNA配列ライブラリからデータを読み出すことは、ユニークなDNA分子の配列を決定し、配列情報を必要に応じて元のデジタルデータに変換することです(図5)。
DNAデータ記憶媒体の利点
前述のように、世界のデータは急激に指数関数的に増加しています。 従来のメディアでは、このような大容量データの保存に対応しきれませんでした。 DNAは、高密度、高複製効率、長期耐久性、長期安定性などの潜在的な利点を持つ、デジタルデータ保存の可能な媒体として機能する可能性があります(https:// www.scientificworldinfo.com)。 DNAの理論的な最大容量は、1ヌクレオチドあたり約2ビットをコード化することができます。 2011年にIBMが建設したデータセンター全体では、約100ペタバイト(PBs)のデータ保存容量があります。 しかし、DNAは高密度であるため、小さなサイズで大量のデータを保存することができます。 1グラムのDNAは理論上最大で約200PBsのデータを保存でき、これはIBMのデータセンター全体のほぼ2倍に相当する。 言い換えれば、世界中で記録されたすべての情報は、数キログラムの DNA に格納することができます。これは、従来のメディアには何百万もの大規模なデータ ストレージ センターが必要であるのに対し、靴箱 1 つ分にしか相当しません。
データ化された DNA メディアは、その高い耐久性により、長期保存が可能です。 さらに悪い環境下でも、DNA の半減期は数百年に達します。 DNAは低温でも高温でも安定しており、その温度範囲は-800℃から800℃までと広い。 また、DNAメディアは、従来のデジタルデータメディアよりもデータの安全性を高めることができる。 新しいデータは指数関数的に増加していますが、そのほとんどは長期保存のためにアーカイブに保存されています。 このようなコールドデータは、すぐに取り出したり、頻繁に使用したりすることはないでしょう。 そのため、DNAメディアへの保存は、簡単で便利、かつ低コストで実現できます。 もう一つの利点は、DNAが高度に保存されていることです。 天然のDNAは、高い効率で、常に塩基対規則(AとT、CとG)に従って正確に自己複製することができます(図3)。
DNAデータストレージメディアの課題
そのユニークな特性に基づいて、従来のメディアと比較して、DNAはデジタルデータストレージのための潜在的かつ有望なメディアである可能性があります。 しかし、DNAが商業的に応用されるには、まだ長い道のりがあります。
データ ストレージにおける DNA の使用は、テープ、ディスク、HDD (ハード ディスク ドライブ) などの従来のメディアよりもはるかに高価です (https://www.scientificworldinfo.com)。 現在、データのエンコードとデコードには、1メガバイト(MB)あたり15,000ドル近いコストがかかっています。 一方、現在のDNA合成技術には限界があり、短いオリゴDNA配列しか合成することができません。 1つのオリゴDNA断片の最大長は数百ヌクレオチドが限界である。 そのため、1つのアーカイブファイルを保存するために、特に1つの大きなファイルでは、数十万個のオリゴDNAが必要になることがあります。 また、オリゴDNAへの書き込み、取り出しには、データのバイナリ化、オリゴDNAへのエンコード、DNA配列の合成・保存、DNA保存ライブラリからの固有配列の取り出し、配列決定・デコード、バイナリからデータへの変換など、複数のステップを必要とし、時間がかかるとされています。 ディスクやテープなどの従来のメディアは論理的なアドレス情報を持っていますが、オリゴDNAは持っていません。 したがって、我々が期待するユニークなコード化されたDNA配列のアドレス指定は非常に困難です。 一方、DNAベースのデータストレージでは、ランダムアクセスが重要ですが、オリゴDNAはランダムアクセス能力を持ちません。 現在のところ、DNAデータストレージでは、バルクアクセスしかできません。 そのため、1バイトを読み取るだけであっても、DNAデータストレージ全体をソートし、配列決定し、デコードしなければならない。 そのため、正しいDNA配列を選択的に取り出すための適切なプライマーが必要とされる。 また、このプライマーは、DNA配列の解読やデータの取り出しの際に、ランダムアクセスを可能にする。 ユニークなプライマーを用いたシーケンシングは、DNAライブラリー全体ではなく、必要なオリゴDNAだけを選択的に読み取ることができる。 また、現在、DNAの合成や塩基配列の決定は、完全ではありません。 DNA合成や塩基配列決定の際には、挿入、欠失、置換などのエラーが発生する可能性があり、そのエラー率は1塩基あたり約1%である。
DNA データストレージ媒体
グローバルデータの急激な増加、十分なストレージスペースの不足、革新的なストレージアプローチの要求により、ビッグデータストレージの分野で、潜在的な全く新しい媒体としての DNA が話題になっています。 高密度、高複製効率、長期耐久性、安定性など、DNAは従来のデータ記憶媒体を凌駕する独自の利点を備えています。 しかし、DNAは高価であること、ランダムアクセスができないこと、データのエンコードやデコードに時間がかかることなどから、デジタルデータストレージへの応用は限定的でした。 しかし、DNA技術の進歩は目覚しいものがあります。 例えば、最初のヒトゲノムの塩基配列を決定するために、世界中の科学者が約10年から20年の歳月をかけて協力し、2013年には総費用が30億ドルに達しました(ヒトゲノム計画(HGP)のウェブサイト。 https://www.genome.gov/human-genomeproject)
まとめ
現在では、科学者は数千ドルと数週間あれば、1つのヒトゲノム全体の配列を完成させることができるようになりました。 そして、近い将来、1つのヒトゲノムの配列決定には、数時間で100ドル以下しかかからないと予想されます。 このように、コスト面でもアフォーダブルになることが期待されています。 ランダムアクセスとアドレス指定については、科学者たちが独自のプライマーを設計し、必要な情報を選択的にアドレス指定し、取り出すことでこの課題を解決してきました。 また、エラー発生を防ぐために、エラー訂正用メタデータをオリゴDNA断片にコード化している。 一方、1分子DNAシーケンサーが発明され、現在では入手可能である。 1分子DNAシーケンサーは、ハンディで持ち運びが便利である。 1分子DNAシーケンサーは、携帯に便利で、DNAシーケンシングのコストを下げ、DNA情報の検索を容易にすることができる。 したがって、DNAデータストレージの技術の進歩に続いて、データストレージ媒体として機能するDNAは、このビッグデータの時代における黄金の機会になるでしょう
- Mayer C, McInroy GR, Murat P, Delft PV, Balasubramanian S (2016) An epigenetics-inspired DNA-based data storage system. Angew Chem Int Ed Engl 55: 11144-11148.
- Swati A, Mathuria, F, Bhavani, S, Malathy E, Mahadevan R (2017) A review on various encoding schemes used in digital DNA data storage.「デジタルDNAデータストレージで使用される様々なエンコーディングスキームのレビュー」。 Int J Civil Eng Technol 8: 7-10.
- Appuswamy RLK, Barbry P, Antonini M, Madderson O, Freemont P (2019) アーカイブ: DBMSのストレージ階層でDNAを利用する。 CIDR 2019, Biennal Conference on Innovative Data Systems Research, California, USA.
- De Silva PY, GU Ganegoda (2016) New trends of digital data storage in DNA. Biomed Res Int pp: 8072463-8072472.
- Panda DM, Baig KA, Swain MJ, Behera A, Dash D (2018) DNA as a digital information storage device: hope or hype? Biotech 8: 9-15.
- Chen K, Kong J, Zhu J, Ermann N, Predki P, et al. (2019) DNAナノ構造および固体ナノポアを用いたデジタルデータストレージ. Nano Lett 19: 1210-1215.
- Yazdi S, Gabrys R, Milenkovic O (2017) Portable and error-free DNA-based data storage.(英語).(英語).(英語).(英語).(英語).(英語).(英語).(英語).(英語).(英語). Sci Rep 7: 5011-5013.
- Church GM, Gao Y, Kosuri S (2012) 次世代デジタル情報ストレージ in DNA. Science 337: 1628-1630.
- Kuang SY, Zhu G, Wang ZL (2018) Triboelectrification-Enabled Self-Powered Data Storage. Adv Sci (Weinh) 5: 1700658.
- Block FE (1987) Analog and digital computer theory. Int J Clin Monit Comput 4: 47-51.
- O’ Driscoll A, Sleator RD (2013) Synthetic DNA: the next generation of big data storage. Bioengineered 4: 123-1235.
- Portin P (2014) The birth and development of the DNA theory of inheritance: sixty years since the discovery of the structure of DNA.(DNAの構造の発見から60年). J Genet 93: 293-302.
- Leu K, Obermayer B, Rajamani S, Gerland U, Chen IA (2011) The prebiotic evolutionary advantage of transferring genetic information from RNA to DNA.J Genet 93: 293-302.
- Portin P (2014) Birth and Development: 60 years from the discovery of DNA structure. Nucleic Acids Res 39: 8135-8147.
- Burgers PMJ, Kunkel TA (2017) Eukaryotic DNA replication Fork.(英語)。 Annu Rev Biochem 86: 417-438.
- Akram F, Haq I, Ali H, Laghari AT (2018) Trends to store digital data in DNA: an overview. Mol Biol Rep 45: 1479-1490.
- Organick L, Ang SD, Chen YJ, Lopez R, Yekhanin S, et al. (2018) Random access in large-scale DNA data storage.をご参照ください。 Nat Biotechnol 36: 242-248.
- Bornholt J, Lopez R, Carmean DM, Ceze L, Seelig G, et al. (2016) A DNA-based archival storage system.(英語): A DNA-based archival storage system. ASPLOS 201 (21st ACM International Conference on Architectural Support for Programming Languages and Operating Systems, Atlanta, GA).
- Newman S, Stephenson AP, Willsey M, Nguyen BH, Takahashi CN, et al. (2019) High density DNA data storage library via dehydration with digital microfluidic retrieval.DHN. Nat Commun 10: 1706-1710.
- Yazdi SM, Yuan Y, Ma J, Zhao H, Milenkovic O (2015) A rewritable, random-access DNA-based storage system.(英語)。 Sci Rep 5: 14138-14140.
- Ahn T, Ban H, Park H (2018) ロングリードDNAにデジタル情報を保存する。 Genomics Inform 16: e30-35.
- Bayley H (2017) Single-molecule DNA sequencing: 井戸の底に到達する。 Nat Nanotechnol 12: 1116-1117.
.