Health Science Journal
Nyckelord
Digital datalagring; Desoxyribonukleinsyra (DNA); Binär; Kodning; Avkodning; Sekvensering
Introduktion
Nuförtiden är det en explosiv era av stora data. Dessa stora data finns och täcker nästan överallt, från livsmedelsbutiker till banker, från offline till online, från akademi till industri, från sjukhus till samhälle, från organisation till regering. Lagring och hantering av stora data börjar bli ett allvarligt problem. För närvarande lagras de flesta data världen över huvudsakligen på magnetiska och optiska medier, t.ex. hårddiskar, skivor, cd-skivor, band, dvd-skivor, bärbara hårddiskar och USB-enheter. Den ökande hastigheten för dessa arkivdata ökar dock explosionsartat i exponentiell takt. Dessa traditionella medier och deras begränsade kapacitet för datalagring kan inte uppfylla kraven för den snabba ökningen av digitala data. Samtidigt är dessa mediers hållbarhet för datalagring en stor utmaning. Deras hållbarhet är mycket begränsad. Dessa medier räcker bara under en mycket begränsad tid. Skivor kan till exempel hålla i flera år och band i flera decennier. Andra elektroniska lagringsenheter kan hållas i gott skick i flera decennier. Kapaciteten för datalagring är ett annat problem när det gäller lagring av stora digitala data. En cd-skiva kan lagra flera hundra megabyte (MB) data. En stor hårddisk kan lagra några terabyte (TB) data. Deras kapacitet är dock långt ifrån de krav som ställs på explosiva informationsdata.
Som Patrizio säger finns det totalt 33 Zettabyte (ZB) data i världen 2018 (https://www.networkworld.com), vilket motsvarar 22 triljoner gigabyte (GB). Därför behövs en ny lagringsteknik och ett innovativt system för att uppfylla kraven i denna moderna tid. Desoxyribonukleinsyra (DNA) förväntas på grund av sina unika fördelar vara ett idealiskt medium för digital datalagring. Att lagra digitala data i DNA är ingen nyhet. Det beskrevs faktiskt av den sovjetiske fysikern Mikhail Neiman på 1960-talet (https:// www.geneticsdigest.com). Det var dock 1988 som man för första gången visade att DNA kan lagra digitala data. Här presenterar vi först tillämpningar av DNA som ett nytt medium för digital datalagring och kommer därefter att diskutera fler detaljer inom detta område där DNA fungerar som datalagringsmedium.
Översyn av tidigare studier
Det binära numeriska systemet
Datorer och andra digitala elektroniska apparater lagrar data och fungerar med det binära numeriska systemet som endast använder två digitala tal eller 0 och 1 . Texterna omvandlas till binär version i datorsystemet. Datorer arbetar och räknar i sin tur med binära siffror och omvandlar så småningom information till läsbara texter. En byte innehåller åtta bitar som består av antingen 0 eller 1 och har 28 (256) möjliga värden (från 0 till 255) och lagrar en enda bokstav (figur 1 och tabell 1) . Som framgår av ASCII-omvandlingstabellen (tabell 1). De tjugosex bokstäverna med stora och små bokstäver konverteras mellan bokstav, binärt och hexadecimalt. För att lagra en stor fil eller ett stort dokument behövs mycket mer minnesdata. En vanlig låt kan behöva dussintals megabyte, med några gigabyte för att lagra en film och flera terabyte för de böcker som lagras i ett stort bibliotek. Som framgår av tabell 2 är mät- och minnesstorlekarna för användning av det binära systemet från den minsta enheten ”byte” till de stora enheterna, inklusive byte (B), kilobyte (KB), megabyte (MB) och gigabyte (GB), terabyte (TB), pegabyte (PB), exabyte (EB), zettabyte (ZB), yottabyte (YB), brontobyte (BB), geopbyte (GPB) och så vidare (https://www.geeksforgeeks.org&https://whatsabyte.com). Enheter som brontobyte (BB), Geopbyte (GPB) är ofattbart stora värden som kanske aldrig kommer att användas i vår verkliga värld (tabell 2).

Figur 1: Textsträngen ”DNA digital data storage” konverterades som binära bitar.
Table 1: The conversion ASCII table of the twenty-six letters with the upper and lower cases among letter, binary and hexadecimal.
| Letter | Binary | Hexadecimal | Letter | Binary | Hexadecimal |
|---|---|---|---|---|---|
| A | 1000001 | 41 | a | 1100001 | 61 |
| B | 1000010 | 42 | b | 1100010 | 62 |
| C | 1000011 | 43 | c | 1100011 | 63 |
| D | 1000100 | 44 | d | 1100100 | 64 |
| E | 1000101 | 45 | e | 1100101 | 65 |
| F | 1000110 | 46 | f | 1100110 | 66 |
| G | 1000111 | 47 | g | 1100111 | 67 |
| H | 1001000 | 48 | h | 1101000 | 68 |
| I | 1001001 | 49 | i | 1101001 | 69 |
| J | 1001010 | 4A | j | 1101010 | 6A |
| K | 1001011 | 4B | k | 1101011 | 6B |
| L | 1001100 | 4C | l | 1101100 | 6C |
| M | 1001101 | 4D | m | 1101101 | 6D |
| N | 1001110 | 4E | n | 1101110 | 6E |
| O | 1001111 | 4F | o | 1101111 | 6F |
| P | 1010000 | 50 | p | 1110000 | 70 |
| Q | 1010001 | 51 | q | 1110001 | 71 |
| R | 1010010 | 52 | r | 1110010 | 72 |
| S | 1010011 | 53 | s | 1110011 | 73 |
| T | 1010100 | 54 | t | 1110100 | 74 |
| U | 1010101 | 55 | u | 1110101 | 75 |
| V | 1010110 | 56 | v | 1110110 | 76 |
| W | 1010111 | 57 | w | 1110111 | 77 |
| X | 1011000 | 58 | x | 1111000 | 78 |
| Y | 1011001 | 59 | y | 1111001 | 79 |
| Z | 1011010 | 5A | z | 1111010 | 7A |
*Note: ACSII (American Standard Code for Information Interchange): serial digital codes to represent number, letters, numerals, and other symbols and to be used as a standard format in the computer system.
Table 2: The sizes of measurement and memory.
| Sizes | Byte Magnitude | Units | Storage* |
|---|---|---|---|
| 1 B | 100 | Byte | A character ”A”, ”1”, ”$” |
| 10 B | 101 | ||
| 100 B | 102 | ||
| 1 KB | 103 | Kilo byte | The size for graphics of small websites ranges between 5 and 100 KB |
| 10 KB | 104 | ||
| 100 KB | 105 | ||
| 1 MB | 106 | Mega byte ( 1 MB: 1 million) |
The size for a high resolution JPEG image is about 1-5 MB |
| 10 MB | 107 | The size for a 3-minute song is about 30 MB | |
| 100 MB | 108 | ||
| 1 GB | 109 | Giga byte | The size for a standard DVD drive is about 5 GB |
| 10 GB | 1010 | (1 GB: 1 billion) | |
| 100 GB | 1011 | ||
| 1 TB | 1012 | Tera byte (1 TB: 1 trillion) |
The size for a typical internal HDD is about 2 TB |
| 10 TB | 1013 | ||
| 100 TB | 1014 | ||
| 1 PB | 1015 | Peta byte (1 PB: 1 quadrillion) |
Google store over 100 PB of all data in their drivers. |
| 10 PB | 1016 | ||
| 100 PB | 1017 | ||
| 1 EB | 1018 | Exa byte (1 EB: 1 quintillion) |
Several hundred EBs of data are transferred over global internet per year Facebook built an entire data center to store 1 EB of data in 2013 |
| 10 EB | 1019 | ||
| 100 EB | 1020 | ||
| 1 ZB | 1021 | Zetta byte (1 ZB: 1 sextillion) |
33 ZBs of global data in 2018. 160-180 ZBs of data is predicted in 2025. |
| 10 ZB | 1022 | ||
| 100 ZB | 1023 | ||
| 1 YB | 1024 | Yotta byte (1 YB: 1 septillion) |
1YB = 1 million EBs 1 YB = Size of the entire World Wide Web |
| 10 YB | 1025 | ||
| 100 YB | 1026 | ||
| 1 BB | 1027 | Bronto byte (1 BB: 1 octillion) |
1BB equals to 1 million ZBs The only thing there is to say about a Brontobyte is that it is a 1 followed by 27 zeros! |
| 10 BB | 1028 | ||
| 100 BB | 1029 | ||
| 1 GPB | 1030 | Geop byte (1 GPB: 1 nonillion) | 1, No one knows why this term was created. It is highly doubtful that anyone alive today will EVER see a Geopbyte hard drive. |
The digital data storage
Digital Data Storage (DDS) was introduced and developed in 1980s. It is a computer-based data storage technology that is based on the Digital Audio Tape (DAT) format. These digital data were stored on the silicon-based chips. Silicon is the primary material of most semiconductor and microelectronic chips. Rent kisel av minneskvalitet finns sällan i naturen. All kisel av mikrochipkvalitet i världen förväntas ta slut inom en snar framtid. Moores lag (antalet transistorer i de integrerade kretsarna fördubblas nästan vartannat år, eller fler transistorer som gör att chipen går snabbare med fler transistorer) håller också på att ta slut. Chipen kan alltså inte rymma fler transistorer och kommer att nå gränsen för sin kapacitet.
Under tiden lagras de flesta digitala data på traditionella magnetiska, optiska och andra medier, t.ex. hårddiskar (HDD, hard disk drive) och CD-skivor. Förutom den begränsade datalagringskapaciteten kan dessa medier också sparas under en mycket begränsad tid. De är känsliga för miljön eller för datalagringsförhållanden. Alla förändringar i miljön och i förhållandena, t.ex. magnetisk exponering, hög fuktighet, hög temperatur och mekanisk skada, kan leda till att medierna skadas eller att data går förlorade. Frekvent användning kan också leda till att de skadas eller att data går förlorade. För att lagra stora datamängder och för att uppfylla kraven på en explosiv ökning av datamängderna behöver vi en stor mängd medier som diskar, CD, DVP och hårddiskar. Detta leder till höga kostnader och är tidskrävande.
Samtidigt ökar ökningen av digitala data och behovet av datalagring i exponentiell takt. IBM byggde 2011 ett stort center med en datalagringskapacitet på 120 PB. Facebook byggde ett annat större center med kapacitet att lagra 1 EB (1000 PBs) data 2013. Alla digitala användare världen över producerade över 44 exabyte (EB) (44000 PB) data per dag. Det producerades totalt 1 zettabyte (ZB, 1000 EBs eller 1 miljon PBs) data globalt 2010 och 33 zettabytes (ZB) data 2018, och 150-200 zettabytes (ZBs) förutspås 2025 (citerat från Datanamis webbplats: https:// www.datanami.com och Network world webbplats: https:// www.networkworld.com) . För att lagra dessa data skulle det behövas hundratusentals enorma rymdcentraler. Under 2018 hade Facebook sammanlagt 15 datacenterplatser under 2018, och fler nya centra har tillkännagivits. De kommer att bygga ytterligare fyra datacenter i Nebraska, som består av sex stora byggnader med datalagringsutrymme på över 2,6 miljoner kvadratmeter. Hur som helst kan datastoringsutrymmet aldrig fånga upp den exponentiella ökningen av data. De nuvarande lagringsmedlen kan inte heller tillgodose lagringsbehovet. Det finns ett brådskande behov av att utveckla en ny generation teknik för datalagring i stället för den nuvarande kiselbaserade datalagringen. Med sina unika egenskaper och potentiella fördelar är desoxyribonukleinsyra (DNA) som ett möjligt digitalt datalagringsmedium på väg in i datalagringens centrum.
Den grundläggande informationen om desoxyribonukleinsyra (DNA)
Å 1953 avslöjade Dr. Crick och Watson att en DNA-molekyl har dubbla strängar som ringlar sig runt varandra och bildar en dubbelspiralformad struktur . Generellt sett är det genetiska materialet i de flesta naturliga organismer dubbelsträngar av spiralformade DNA, varav vissa är enkelsträngar av DNA och andra är enkel- eller dubbelsträngar av RNA. DNA-komponenter eller nukleotider består av kvävebaser, fosfatgrupper och desoxyribose-grupper. De två bokstäverna är strukturerade som ryggraden i varje DNA-molekyl, där varje baspar från varje sträng förbinds genom en vätebindning. DNA-nukleotider består av fyra typer av baser, däribland adenin (A), cytosin (C), guanin (G) och tymin (T) (figur 2) , medan ribonukleinsyra (RNA) har fyra typer av baser, däribland adenin (A), cytosin (C), guanin (G) och uracil (U) i stället för tymin (T). Adenin (A) och guanin (G) är purin, medan cytosin (C), tymin (T) och uracil (U) är pyrimidin . In DNA molecules, the base-pairing rule is that A pairs with T, and G pairs with C (Figures 3 and 4) .
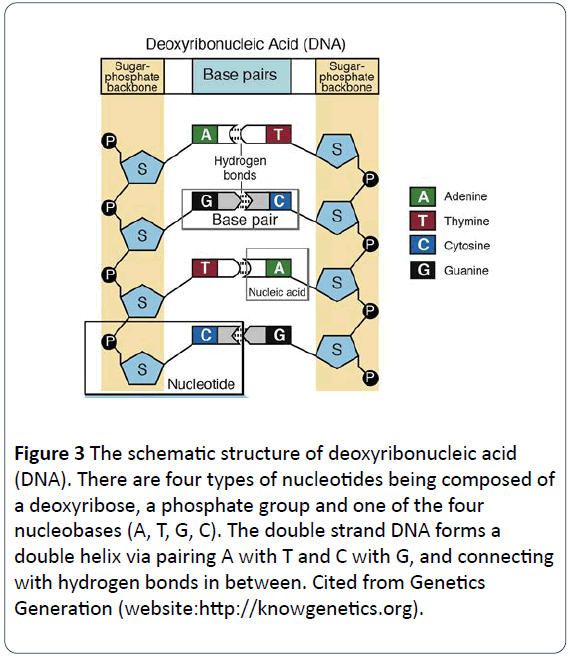
Figure 3: The schematic structure of deoxyribonucleic acid (DNA). There are four types of nucleotides being composed of a deoxyribose, a phosphate group and one of the four nucleobases (A, T, G, C). The double strand DNA forms a double helix via pairing A with T and C with G, and connecting with hydrogen bonds in between. Cited from Genetics Generation (website:https://knowgenetics.org).
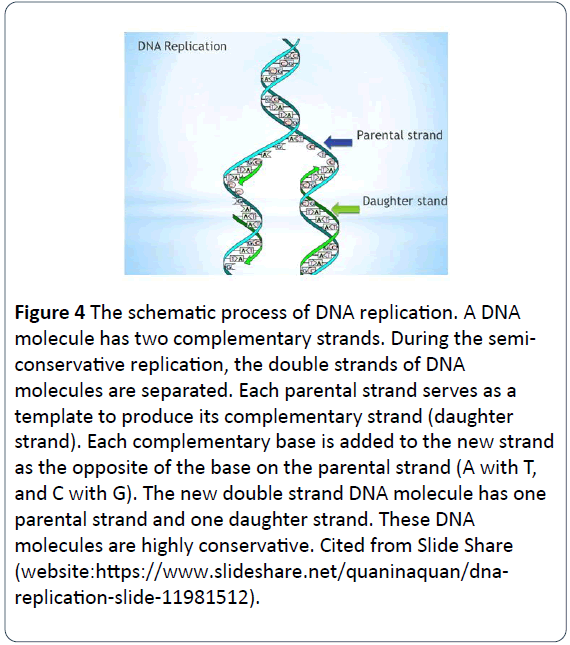
Figure 4: Den schematiska processen för DNA-replikation. En DNA-molekyl har två komplementära strängar. Under den semikonservativa replikationen separeras DNA-molekylernas dubbelsträngar. Varje föräldrasträng tjänar som mall för att producera sin komplementära sträng (dottersträng). Varje komplementär bas läggs till den nya strängen som motsatsen till basen på föräldrasträngen (A med T och C med G). Den nya dubbelsträngade DNA-molekylen har en föräldrasträng och en dottersträng. Dessa DNA-molekyler är mycket konservativa. Citerat från Slide Share (webbplats:https://www.slideshare.net/quaninaquan/dnareplication- slide-11981512).
Processen är att en dubbelsträngad DNA-molekyl avvecklas med var och en av de två föräldrasträngarna som separeras och fungerar som en föräldramall för syntesen av nya dotter-DNA-molekyler. De komplementära nukleotiderna läggs till i dottersträngen, med fosfater och deoxyriboser för att bilda ryggraden för de nya nukleotiderna och nya baser för att para ihop sig med motsatsen till baserna på föräldrasträngen via basparningsregeln (A parar sig med T och G parar sig med C) och för att hålla sig på plats med vätebindningar . Så småningom har varje ny dubbelsträngad DNA-molekyl en föräldrasträng och en dottersträng. DNA-molekylerna replikerar sig i denna halvkonservativa modell, håller genetiska DNA konservativa och konstanta och överförs från en generation till en annan generation (figur 5) .
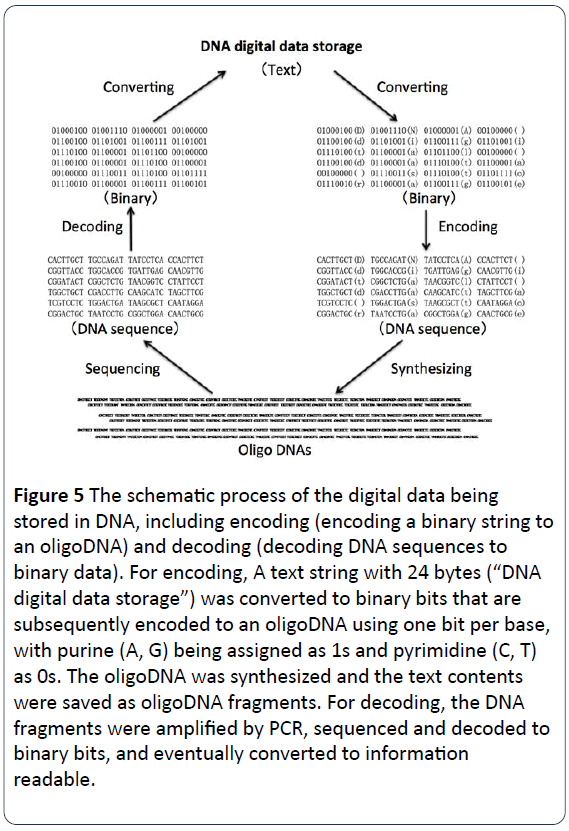
Figur 5: Den schematiskt beskrivna processen för att lagra digitala data i DNA, inklusive kodning (kodning av en binär sträng till ett oligoDNA) och avkodning (avkodning av DNA-sekvenser till binära data). För kodning omvandlades en textsträng med 24 bytes (”DNA digital data storage”) till binära bitar som sedan kodas till ett oligoDNA med en bit per bas, där purin (A, G) tilldelas som 1:or och pyrimidin (C, T) som 0:or. OligoDNA syntetiserades och textinnehållet sparades som oligoDNA-fragment. För avkodning amplifierades DNA-fragmenten med PCR, sekvenserades och avkodades till binära bitar och omvandlades slutligen till läsbar information.
Processen för digital datalagring av DNA
Processen för digital datalagring av DNA går ut på att koda och avkoda binära data till och från syntetiserade DNA-strängar. Texter, siffror, bilder och annat som är läsbart eller synligt omvandlas först till binära språk med 0 och 1 i stället, och kodas sedan till DNA-nukleotidsekvenser, med de fyra baserna (A, C, G, T) i stället för 0 och 1 . Exempelvis är en stor bokstav ”D” ”01000100” på binärt språk, en liten bokstav ”d” är ”01100100” med ett tomt ” ” är ”00100000” (figur 1). I figurerna 3 och 5 har meningen ”DNA digital datalagring” konverterats till binär version för att erhålla binära koder med 24 byte. Därefter kodas binära koder (binära bitar) till DNA-koder. Var och en av de fyra baserna (A, C, G och T) ska tilldelas antingen 1 eller 0. Purin (A, G) tilldelas till exempel 1, medan pyrimidin (C, T) tilldelas 0. Eller så tilldelas de två baserna G och T 1s och de andra två baserna A och C 0s. Som visas i figur 4, för datakodning, omvandlades en textsträng med 24 bytes (”DNA digital data storage”) till binära bitar som sedan kodas till ett oligo-DNA med en bit per bas, där purin (A, G) tilldelas 1 och pyrimidin (C, T) 0. Oligo-DNA syntetiserades kemiskt och textinnehållet sparades sedan som oligo-DNA-fragment för långtidslagring. För datakodning amplifierades DNA-fragmenten med PCR, sekvenserades och avkodades till binära bitar en gång om dagen, vi måste hämta data för att kunna ge ut binära data som är läsbara. Slutligen är läsning av data från DNA-sekvensbibliotek att sekvensera de unika DNA-molekylerna, omvandla sekvenseringsinformationen till ursprungliga digitala data enligt behov eller krav (figur 5) .
Fördelarna med DNA-datalagringsmedium
Som nämnts ovan ökar de globala datamängderna kraftigt i exponentiell takt. De traditionella medierna kan inte i tillräcklig utsträckning hantera kravet på stor datalagring. DNA kan fungera som ett möjligt medium för digital datalagring, med sina potentiella fördelar som hög densitet, hög replikeringseffektivitet, långvarig hållbarhet och långsiktig stabilitet (https:// www.scientificworldinfo.com) . DNA kan vid sin teoretiska maximala kapacitet koda ungefär två bitar per nukleotid . Ett helt datacenter som byggdes av IBM 2011 har cirka 100 petabyte (PB) kapacitet för datalagring. På grund av sin höga densitet kan DNA som datalagringsmedium lagra en stor mängd data på en liten yta. Ett enda gram DNA kan teoretiskt sett lagra cirka 200 PBs data, vilket är nästan dubbelt så mycket som hela IBM:s datacenter. Med andra ord kan all information som spelats in över hela världen lagras i flera kilo DNA, eller motsvarande endast en skokartong jämfört med kravet på miljontals stora datalagringscenter för traditionella medier .
Datakodade DNA-medier kan lagras på lång sikt tack vare sin höga hållbarhet . DNA kan hålla i tusentals år på kalla, torra och mörka platser. Även i sämre miljöer är DNA:s halveringstid upp till hundra år. DNA kan förbli stabilt vid låg eller hög temperatur, med ett brett intervall från -800°C till 800°C . DNA-medier kan också säkra data bättre än traditionella digitala datamedier. Även om nya data ökar exponentiellt, sparas de flesta av dem i arkiv för långtidsförvaring. Dessa kalla data kommer inte att hämtas omedelbart eller användas ofta. Att lagra dem i DNA-media är därför enkelt, bekvämt och kostnadsfritt. En annan fördel är att DNA är mycket välbevarat. De naturliga DNA:erna kan replikera sig själva med hög effektivitet och alltid med basparningsregeln (A med T, C med G) (figur 3) . DNA-mediet kan således i hög grad bevara datatrogenheten under lång tid.
Utmaningarna för DNA-mediet för datalagring
Baserat på dess unika egenskaper och jämfört med de traditionella medierna skulle DNA kunna vara ett potentiellt och lovande medium för digital datalagring. Det är dock fortfarande en lång väg att gå innan DNA kan användas kommersiellt. De utmaningar som vi måste ta itu med finns i olika aspekter, bland annat hög kostnad, låg genomströmning, begränsad tillgång till datalagring, korta syntetiska oligo-DNA-fragment, felfrekvens vid syntes och sekvensering.
Användningen av DNA för datalagring är mycket dyrare än de andra traditionella medierna som band, disk och HDD (hårddisk) (https://www.scientificworldinfo.com) . För närvarande kostar det nästan 15 000 dollar per megabyte (MB) att koda och avkoda data. Samtidigt är den nuvarande tekniken för DNA-syntes begränsad, och endast korta oligodNA-sekvenser kan syntetiseras. Den maximala längden på varje oligo-DNA-fragment är begränsad till flera hundra nukleotider . För att lagra en enda arkiverad fil, särskilt en stor fil, kan det således krävas hundratusentals oligo-DNAs. Det är också tidskrävande att skriva in data i och hämta data från oligo-DNA, eftersom det krävs flera steg, bl.a. omvandling av data till binärdata, kodning av binärdata till oligo-DNA, syntetisering och lagring av DNA-sekvenser, hämtning av unika sekvenser från DNA-lagringsbiblioteket, sekvensering och avkodning och slutligen omvandling av binärdata till dataläsbar data. De traditionella medierna, t.ex. diskar och band, har sin logiska adresseringsinformation, men det har inte oligodNA. Det är därför mycket svårt att adressera den unika kodade DNA-sekvensen som vi förväntar oss att ha . Samtidigt är det viktigt med slumpmässig åtkomst till DNA-baserad datalagring, men oligodNA har ingen förmåga till slumpmässig åtkomst. Med nuvarande metoder kan DNA-datalagring endast ske i stor skala. Hela det DNA-baserade datalagret måste sorteras, sekvenseras och avkodas från DNA-datalagret även om vi bara behöver läsa en enda byte . Därför krävs rätt primer som används för att selektivt hämta rätt DNA-sekvens. Detta kommer också att ge en slumpmässig åtkomst under DNA-sekvensering och datahämtning. Sekvenseringen med den unika primern kan selektivt läsa endast det oligoDNA som krävs, snarare än hela DNA-biblioteket . För närvarande är DNA-syntes och sekvensering inte helt perfekta. Under DNA-syntesen och sekvenseringen kan det förekomma inlagring, borttagning, substitution och andra fel, med en felfrekvens på cirka 1 % per nukleotid . Tekniken och kostnaden för DNA-syntes och sekvensering är inte lämpliga för nuvarande datalagring.
Respektive för DNA som datalagringsmedium
På grund av den exponentiella ökningen av globala data, bristen på tillräckliga lagringsutrymmen och kravet på innovativa lagringsmetoder blir DNA som ett potentiellt helt nytt medium ett hett ämne inom området för lagring av stora datamängder. Med hög densitet, hög replikeringseffektivitet, långsiktig hållbarhet och stabilitet uppvisar DNA sina egna fördelar jämfört med traditionella datalagringsmedier . Samtidigt har tillämpningarna av digital datalagring med DNA varit begränsade på grund av den höga kostnaden, bristande förmåga till slumpmässig åtkomst och tidskrävande kodning och avkodning av data. Lyckligtvis går framstegen inom DNA-tekniken snabbt framåt. För att slutföra sekvenseringen av det första mänskliga genomet samarbetade och arbetade forskare världen över i cirka 10-20 år, med en total kostnad på 3 miljarder dollar 2013 (webbplatsen Human Genome Project (HGP)): https://www.genome.gov/human-genomeproject).
Slutsats
Nuförtiden behöver forskarna bara några tusen dollar och några veckor för att slutföra sekvensen av ett helt mänskligt genom. Och det förväntas att sekvenseringen av ett mänskligt genom bara kommer att kosta hundra dollar eller mindre under flera timmar inom en nära framtid. Kostnaden kan alltså förväntas vara överkomlig. När det gäller slumpmässig åtkomst och adressering av information har forskarna löst denna utmaning genom att utforma unika primers för att selektivt adressera och hämta den information som krävs. För att undvika att fel uppstår kodas metadata för felkorrigering i oligo-DNA-fragment. Under tiden har DNA-sekvenser för enstaka molekyler uppfunnits och finns för närvarande tillgängliga. De är behändiga och bärbara. De kan ytterligare minska kostnaderna för DNA-sekvensering och förenkla hämtningen av DNA-information. Efter framstegen inom tekniken för DNA-datalagring kommer således DNA som datalagringsmedium att vara ett gyllene tillfälle i denna era av stora datamängder.
- Mayer C, McInroy GR, Murat P, Delft PV, Balasubramanian S (2016) An epigenetics-inspired DNA-based data storage system. Angew Chem Int Ed Engl 55: 11144-11148.
- Swati A, Mathuria, F, Bhavani, S, Malathy E, Mahadevan R (2017) A review on various encoding schemes used in digital DNA data storage. Int J Civil Eng Technol 8: 7-10.
- Appuswamy RLK, Barbry P, Antonini M, Madderson O, Freemont P (2019) Archive: Användning av DNA i DBMS-lagringshierarkin. CIDR 2019, Biennal Conference on Innovative Data Systems Research, California, USA.
- De Silva PY, GU Ganegoda (2016) New trends of digital data storage in DNA. Biomed Res Int pp: 8072463-8072472.
- Panda DM, Baig KA, Swain MJ, Behera A, Dash D (2018) DNA as a digital information storage device: hope or hype? Biotech 8: 9-15.
- Chen K, Kong J, Zhu J, Ermann N, Predki P, et al. (2019) Digital datalagring med hjälp av DNA-nanostrukturer och fasta nanoporer. Nano Lett 19: 1210-1215.
- Yazdi S, Gabrys R, Milenkovic O (2017) Bärbar och felfri DNA-baserad datalagring. Sci Rep 7: 5011-5013.
- Church GM, Gao Y, Kosuri S (2012) Next-generation digital information storage in DNA. Science 337: 1628-1630.
- Kuang SY, Zhu G, Wang ZL (2018) Triboelectrification-Enabled Self-Powered Data Storage. Adv Sci (Weinh) 5: 1700658.
- Block FE (1987) Analog and digital computer theory. Int J Clin Monit Comput 4: 47-51.
- O’ Driscoll A, Sleator RD (2013) Synthetic DNA: the next generation of big data storage. Bioengineered 4: 123-1235.
- Portin P (2014) The birth and development of the DNA theory of inheritance: sixty years since the discovery of the structure of DNA. J Genet 93: 293-302.
- Leu K, Obermayer B, Rajamani S, Gerland U, Chen IA (2011) The prebiotic evolutionary advantage of transfering genetic information from RNA to DNA. Nucleic Acids Res 39: 8135-8147.
- Burgers PMJ, Kunkel TA (2017) Eukaryotic DNA replication Fork. Annu Rev Biochem 86: 417-438.
- Akram F, Haq I, Ali H, Laghari AT (2018) Trends to store digital data in DNA: an overview. Mol Biol Rep 45: 1479-1490.
- Organick L, Ang SD, Chen YJ, Lopez R, Yekhanin S, et al. (2018) Random access in large-scale DNA data storage. Nat Biotechnol 36: 242-248.
- Bornholt J, Lopez R, Carmean DM, Ceze L, Seelig G, et al. (2016) A DNA-based archival storage system. ASPLOS 201 (21st ACM International Conference on Architectural Support for Programming Languages and Operating Systems, Atlanta, GA).
- Newman S, Stephenson AP, Willsey M, Nguyen BH, Takahashi CN, et al. (2019) High density DNA data storage library via dehydration with digital microfluidic retrieval. Nat Commun 10: 1706-1710.
- Yazdi SM, Yuan Y, Ma J, Zhao H, Milenkovic O (2015) A rewritable, random-access DNA-based storage system. Sci Rep 5: 14138-14140.
- Ahn T, Ban H, Park H (2018) Storage digital information in the long read DNA. Genomics Inform 16: e30-35.
- Bayley H (2017) Single-molecule DNA sequencing: Getting to the bottom of the well. Nat Nanotechnol 12: 1116-1117.