Health Science Journal
Słowa kluczowe
Cyfrowe przechowywanie danych; Kwas dezoksyrybonukleinowy (DNA); Binary; Kodowanie; Dekodowanie; Sekwencjonowanie
Wprowadzenie
Teraz jest to wybuchowa era big data. Te big data istnieją i obejmują prawie wszędzie, od sklepów spożywczych do banków, od offline do online, od akademii do przemysłu, od szpitala do społeczności, od organizacji do rządu. Przechowywanie i zarządzanie dużymi danymi staje się poważnym problemem. Obecnie większość danych na świecie jest przechowywana głównie na nośnikach magnetycznych i optycznych, takich jak HDD (hard disk drive), DYSK, CD, taśmy, DVD, przenośne dyski twarde i pamięci USB. Jednak szybkość wzrostu tych danych archiwalnych eksplozywnie wzrasta w tempie wykładniczym. Te tradycyjne nośniki i ich ograniczona pojemność nie są w stanie sprostać wymaganiom szybkiego przyrostu danych cyfrowych. Tymczasem, trwałość przechowywania danych na tych nośnikach jest jednym z głównych wyzwań. Ich trwałość jest bardzo ograniczona. Nośniki te działają tylko przez bardzo ograniczony czas. Dla przykładu, dyski mogą wytrzymać kilka lat, a taśmy kilkadziesiąt lat. Inne elektroniczne pamięci masowe mogą być przechowywane w dobrym stanie przez kilkadziesiąt lat. Pojemność przechowywania danych jest kolejnym problemem przy przechowywaniu dużych danych cyfrowych. Płyta CD może przechowywać kilkaset megabajtów (MB) danych. Duży dysk twardy może przechowywać kilka terabajtów (TB) danych. Jednak ich pojemność jest daleka od wymagań eksplodujących danych informacyjnych .
Jak powiedział Patrizio, w 2018 roku na całym świecie jest 33 Zettabajty (ZBs) danych (https://www.networkworld.com), równe 22 bilionom gigabajtów (GBs). Dlatego, aby sprostać wymaganiom tej nowoczesnej ery, potrzebna jest nowa technologia przechowywania danych i innowacyjny system. Kwas dezoksyrybonukleinowy (DNA), ze względu na swoje unikalne zalety, ma być idealnym medium do przechowywania danych cyfrowych. Przechowywanie danych cyfrowych w DNA nie jest nową historią. Właściwie została ona opisana przez radzieckiego fizyka Michaiła Neimana w latach 60-tych (https:// www.geneticsdigest.com). Jednakże, po raz pierwszy zademonstrowano, że DNA może przechowywać dane cyfrowe w 1988 roku. Tutaj, po pierwsze wprowadzić zastosowania DNA jako nowego medium w cyfrowym przechowywaniu danych, a następnie omówić więcej szczegółów w tej dziedzinie DNA służąc jako dane-storing medium.
Review of Previous Studies
Binarny system numeryczny
Komputery i inne cyfrowe urządzenia elektroniczne przechowywać dane i działać z binarnego systemu numerycznego, który wykorzystuje tylko dwie liczby cyfrowe lub 0 i 1 . Teksty są konwertowane do wersji binarnej w systemie komputerowym. Z kolei komputery działają i obliczają w systemie binarnym, ostatecznie konwertują informacje do tekstów czytelnych. Jeden bajt zawiera osiem bitów składających się z 0 lub 1 i mających 28 (256) możliwych wartości (od 0 do 255), i przechowuje jedną literę (rysunek 1 i tabela 1) . Jak pokazano w tabeli konwersji ASCII (Tabela 1). Dwadzieścia sześć liter z wielkich i małych liter są konwertowane między List, Binary i szesnastkowy. Do przechowywania dużych plików lub dokumentów potrzebują znacznie więcej danych pamięci. Zwykły utwór może potrzebować kilkadziesiąt megabajtów, z kilku gigabajtów do przechowywania filmu i kilka terabajtów dla książek przechowywanych w dużej bibliotece. Jak pokazano w tabeli 2 są rozmiary pomiaru i pamięci dla korzystania z systemu binarnego od najmniejszej jednostki „bajt” do dużych jednostek, w tym bajt (B), kilobajt (KB), megabajt (MB), gigabajt (GB), terabajt (TB), pegabajt (PB), eksabajt (EB), zettabajt (ZB), yottabajt (YB), brontobajt (BB), geopobajt (GPB) i tak dalej (https://www.geeksforgeeks.org&https://whatsabyte.com). Jednostki takie jak brontobajt (BB), geopobajt (GPB) są niewyobrażalnie wielkimi wartościami, które mogą nigdy nie zostać użyte w naszym realnym świecie (Tabela 2).

Rycena 1: Ciąg tekstowy „DNA digital data storage” został przekonwertowany jako binarne bity.
Table 1: The conversion ASCII table of the twenty-six letters with the upper and lower cases among letter, binary and hexadecimal.
| Letter | Binary | Hexadecimal | Letter | Binary | Hexadecimal |
|---|---|---|---|---|---|
| A | 1000001 | 41 | a | 1100001 | 61 |
| B | 1000010 | 42 | b | 1100010 | 62 |
| C | 1000011 | 43 | c | 1100011 | 63 |
| D | 1000100 | 44 | d | 1100100 | 64 |
| E | 1000101 | 45 | e | 1100101 | 65 |
| F | 1000110 | 46 | f | 1100110 | 66 |
| G | 1000111 | 47 | g | 1100111 | 67 |
| H | 1001000 | 48 | h | 1101000 | 68 |
| I | 1001001 | 49 | i | 1101001 | 69 |
| J | 1001010 | 4A | j | 1101010 | 6A |
| K | 1001011 | 4B | k | 1101011 | 6B |
| L | 1001100 | 4C | l | 1101100 | 6C |
| M | 1001101 | 4D | m | 1101101 | 6D |
| N | 1001110 | 4E | n | 1101110 | 6E |
| O | 1001111 | 4F | o | 1101111 | 6F |
| P | 1010000 | 50 | p | 1110000 | 70 |
| Q | 1010001 | 51 | q | 1110001 | 71 |
| R | 1010010 | 52 | r | 1110010 | 72 |
| S | 1010011 | 53 | s | 1110011 | 73 |
| T | 1010100 | 54 | t | 1110100 | 74 |
| U | 1010101 | 55 | u | 1110101 | 75 |
| V | 1010110 | 56 | v | 1110110 | 76 |
| W | 1010111 | 57 | w | 1110111 | 77 |
| X | 1011000 | 58 | x | 1111000 | 78 |
| Y | 1011001 | 59 | y | 1111001 | 79 |
| Z | 1011010 | 5A | z | 1111010 | 7A |
*Note: ACSII (American Standard Code for Information Interchange): serial digital codes to represent number, letters, numerals, and other symbols and to be used as a standard format in the computer system.
Table 2: The sizes of measurement and memory.
| Sizes | Byte Magnitude | Units | Storage* |
|---|---|---|---|
| 1 B | 100 | Byte | A character „A”, „1”, „$” |
| 10 B | 101 | ||
| 100 B | 102 | ||
| 1 KB | 103 | Kilo byte | The size for graphics of small websites ranges between 5 and 100 KB |
| 10 KB | 104 | ||
| 100 KB | 105 | ||
| 1 MB | 106 | Mega byte ( 1 MB: 1 million) |
The size for a high resolution JPEG image is about 1-5 MB |
| 10 MB | 107 | The size for a 3-minute song is about 30 MB | |
| 100 MB | 108 | ||
| 1 GB | 109 | Giga byte | The size for a standard DVD drive is about 5 GB |
| 10 GB | 1010 | (1 GB: 1 billion) | |
| 100 GB | 1011 | ||
| 1 TB | 1012 | Tera byte (1 TB: 1 trillion) |
The size for a typical internal HDD is about 2 TB |
| 10 TB | 1013 | ||
| 100 TB | 1014 | ||
| 1 PB | 1015 | Peta byte (1 PB: 1 quadrillion) |
Google store over 100 PB of all data in their drivers. |
| 10 PB | 1016 | ||
| 100 PB | 1017 | ||
| 1 EB | 1018 | Exa byte (1 EB: 1 quintillion) |
Several hundred EBs of data are transferred over global internet per year Facebook built an entire data center to store 1 EB of data in 2013 |
| 10 EB | 1019 | ||
| 100 EB | 1020 | ||
| 1 ZB | 1021 | Zetta byte (1 ZB: 1 sextillion) |
33 ZBs of global data in 2018. 160-180 ZBs of data is predicted in 2025. |
| 10 ZB | 1022 | ||
| 100 ZB | 1023 | ||
| 1 YB | 1024 | Yotta byte (1 YB: 1 septillion) |
1YB = 1 million EBs 1 YB = Size of the entire World Wide Web |
| 10 YB | 1025 | ||
| 100 YB | 1026 | ||
| 1 BB | 1027 | Bronto byte (1 BB: 1 octillion) |
1BB equals to 1 million ZBs The only thing there is to say about a Brontobyte is that it is a 1 followed by 27 zeros! |
| 10 BB | 1028 | ||
| 100 BB | 1029 | ||
| 1 GPB | 1030 | Geop byte (1 GPB: 1 nonillion) | 1, No one knows why this term was created. It is highly doubtful that anyone alive today will EVER see a Geopbyte hard drive. |
The digital data storage
Digital Data Storage (DDS) was introduced and developed in 1980s. It is a computer-based data storage technology that is based on the Digital Audio Tape (DAT) format. These digital data were stored on the silicon-based chips. Silicon is the primary material of most semiconductor and microelectronic chips. Czysty krzem klasy pamięciowej jest rzadko spotykany w przyrodzie. Przewiduje się, że w najbliższej przyszłości wszystkie zasoby krzemu klasy mikroprocesorowej na świecie zostaną wyczerpane. Również Prawo Moore’a (liczba tranzystorów umieszczonych na układach scalonych jest prawie podwajana co dwa lata, lub chipy działają szybciej dzięki większej liczbie tranzystorów) dobiega końca. W związku z tym chipy nie mogą pomieścić dodatkowych tranzystorów i osiągną limit swojej pojemności.
Większość obecnych danych cyfrowych jest przechowywana na tradycyjnych nośnikach magnetycznych, optycznych i innych, takich jak HDD (hard disk drive) i CD. Oprócz ograniczonej pojemności przechowywania danych, nośniki te mogą być przechowywane przez bardzo ograniczony czas. Są one wrażliwe na środowisko lub warunki przechowywania danych. Wszelkie zmiany środowiskowe i warunkowe, takie jak ekspozycja magnetyczna, wysoka wilgotność, wysoka temperatura, uszkodzenia mechaniczne, mogą spowodować uszkodzenie tych nośników lub utratę danych. Częste użytkowanie może również prowadzić do ich uszkodzenia lub utraty danych. Ponadto, aby przechowywać duże ilości danych i spełnić wymóg gwałtownego wzrostu ilości danych, potrzebujemy dużej ilości takich nośników jak dyski, CD, DVP, dyski twarde. Prowadzi to do wysokich kosztów i jest czasochłonne.
Równocześnie, wzrost ilości danych cyfrowych i wymagania dotyczące przechowywania danych rosną w tempie wykładniczym. W 2011 roku IBM zbudował duże centrum o pojemności 120 PBs. W 2013 roku Facebook zbudował kolejne, większe centrum o pojemności 1 EB (1000 PBs) danych. Wszyscy cyfrowi użytkownicy na całym świecie wytwarzali ponad 44 eksabajty (EB) (44000 PBs) danych dziennie. W 2010 r. na całym świecie wyprodukowano 1 zettabajt (ZB, 1000EB lub 1 milion PBs) danych, w 2018 r. 33 zettabajty (ZB) danych, a w 2025 r. przewiduje się 150-200 zettabajtów (ZB) (cytat ze strony internetowej Datanami: https:// www.datanami.com i strony internetowej Network world: https:// www.networkworld.com) . Do przechowywania tych danych potrzebne byłyby setki tysięcy ogromnych centrów kosmicznych. W 2018 roku Facebook posiadał w sumie 15 lokalizacji centrów danych, przy czym zapowiadane są kolejne nowe centra. Zbudują cztery dodatkowe centra danych w Nebrasce, składające się z sześciu dużych budynków z przestrzenią datastoringową o powierzchni ponad 2,6 miliona stóp kwadratowych. Niezależnie od tego, przestrzeń składowania danych nigdy nie będzie w stanie sprostać wykładniczemu wzrostowi ilości danych. Obecne nośniki pamięci również nie są w stanie zaspokoić zapotrzebowania na przechowywanie danych. Istnieje pilna potrzeba opracowania nowej generacji technologii przechowywania danych zamiast obecnej, opartej na krzemie. Z jego unikalnych cech i potencjalnych zalet, kwas dezoksyrybonukleinowy (DNA) jako możliwe cyfrowe nośniki danych zbliża się do centralnego etapu data storage.
Podstawowe informacje kwasu dezoksyrybonukleinowego (DNA)
W 1953 roku, Dr Crick i Watson ujawnił, że cząsteczka DNA o podwójnych nici, które zwijają się wokół siebie i tworzą podwójną strukturę helikalną. Ogólnie rzecz biorąc, materiały genetyczne w większości organizmów naturalnych to podwójne nici spiralnego DNA, przy czym niektóre z nich są pojedynczymi nićmi DNA, a inne są pojedynczymi lub podwójnymi nićmi RNA. Składniki DNA lub nukleotydy składają się z zasad azotowych, grup fosforanowych i grup deoksyrybozowych. Te dwie litery są zbudowane jako szkielet każdej cząsteczki DNA, z każdej pary zasad z każdej nici, aby połączyć przez wiązanie wodorowe. Nukleotydy DNA składają się z czterech rodzajów zasad, w tym adeniny (A), cytozyny (C), guaniny (G) i tyminy (T) (rys. 2), przy czym kwas rybonukleinowy (RNA) ma cztery rodzaje zasad, w tym adeninę (A), cytozynę (C), guaninę (G) i uracyl (U) zamiast tyminy (T). Adenina (A) i guanina (G) są purynami, a cytozyna (C), tymina (T) i uracyl (U) są pirymidynami. In DNA molecules, the base-pairing rule is that A pairs with T, and G pairs with C (Figures 3 and 4) .
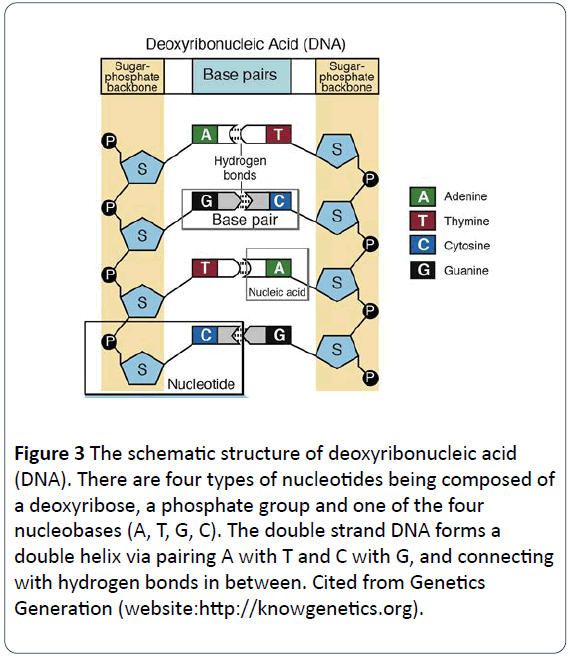
Figure 3: The schematic structure of deoxyribonucleic acid (DNA). There are four types of nucleotides being composed of a deoxyribose, a phosphate group and one of the four nucleobases (A, T, G, C). The double strand DNA forms a double helix via pairing A with T and C with G, and connecting with hydrogen bonds in between. Cited from Genetics Generation (website:https://knowgenetics.org).
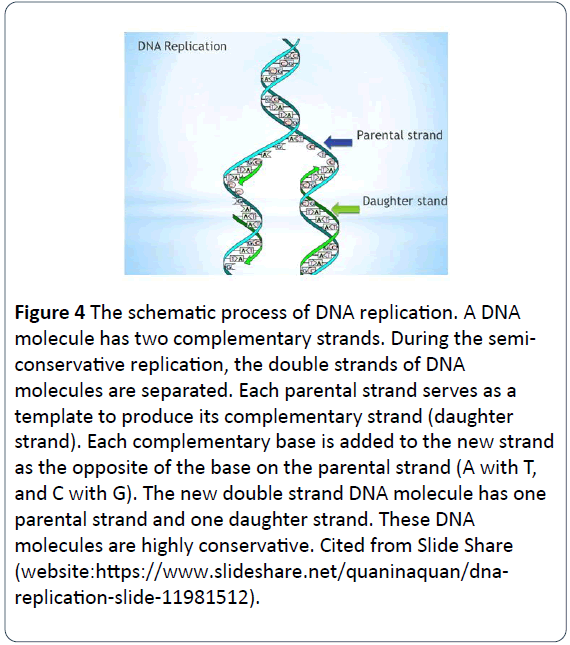
Figure 4: Schematyczny proces replikacji DNA. Cz±steczka DNA posiada dwie komplementarne nici. Podczas semikonserwatywnej replikacji podwójne nici cząsteczek DNA ulegają rozdzieleniu. Każda z nici macierzystych służy jako szablon do wytworzenia swojej nici komplementarnej (nici córki). Każda komplementarna zasada jest dodawana do nowej nici jako przeciwieństwo zasady na nici macierzystej (A z T, a C z G). Nowa dwuniciowa cząsteczka DNA ma jedną nić rodzicielską i jedną nić potomną. Takie cząsteczki DNA są wysoce konserwatywne. Cytat pochodzi z serwisu Slide Share (strona:https://www.slideshare.net/quaninaquan/dnareplication- slide-11981512).
Proces polega na tym, że dwuniciowa cząsteczka DNA odwija się, przy czym każda z dwóch nici rodzicielskich jest rozdzielana i działa jako szablon rodzicielski do syntezy nowych cząsteczek DNA. Komplementarne nukleotydy są dodawane do nici córki, przy czym fosforany i dezoksyrybozy tworzą szkielet nowych nukleotydów, a nowe zasady łączą się w pary z przeciwnymi zasadami na nici rodzicielskiej poprzez zasadę łączenia zasad (A łączy się w pary z T, a G łączy się w pary z C) i utrzymują się na miejscu dzięki wiązaniom wodorowym. Ostatecznie każda z nowych dwuniciowych cząsteczek DNA posiada jedną nić rodzicielską i jedną nić potomną. Cząsteczki DNA replikują się w tym półkonserwatywnym modelu, zachowują konserwatywne i stałe DNA genetyczne i przechodzą z jednego pokolenia na drugie (Rysunek 5) .
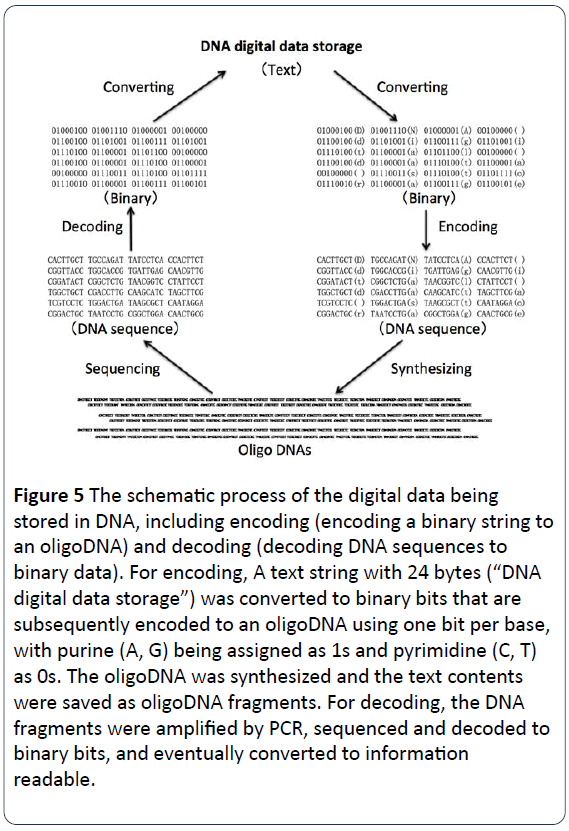
Rysunek 5: Schematyczny proces przechowywania danych cyfrowych w DNA, w tym kodowanie (kodowanie ciągu binarnego do oligoDNA) i dekodowanie (dekodowanie sekwencji DNA do danych binarnych). W przypadku kodowania, ciąg tekstowy o długości 24 bajtów („przechowywanie danych cyfrowych DNA”) przekształcano na bity binarne, które następnie kodowano w oligoDNA przy użyciu jednego bitu na zasadę, przy czym purynie (A, G) przypisywano jako 1s, a pirymidynie (C, T) jako 0s. OligoDNA zostało zsyntetyzowane, a treść tekstu została zapisana jako fragmenty oligoDNA. W celu dekodowania, fragmenty DNA były amplifikowane metodą PCR, sekwencjonowane i dekodowane do bitów binarnych, a ostatecznie przekształcane w informacje możliwe do odczytania.
Proces przechowywania danych cyfrowych DNA
Proces przechowywania danych cyfrowych DNA polega na kodowaniu i dekodowaniu danych binarnych do i ze zsyntetyzowanych nici DNA. Teksty, liczby, obrazy i inne czytelne lub widoczne są najpierw konwertowane do języków binarnych z 0 i 1 zamiast, a następnie kodowane do sekwencji nukleotydów DNA, z czterema zasadami (A, C, G, T) zamiast 0 i 1 . Na przykład, wielka litera „D” to „01000100” w języku binarnym, mała litera „d” to „01100100” z pustym ” ” to „00100000” (rysunek 1). Na rysunkach 3 i 5 zdanie „cyfrowe przechowywanie danych DNA” zostało przekształcone do wersji binarnej, aby uzyskać kody binarne o długości 24 bajtów. Następnie kody binarne (bity binarne) zakodowane są w kodach DNA. Każda z czterech zasad (A, C, G i T) powinna być przypisana jako 1 lub 0. Na przykład puryny (A, G) są przypisane jako 1s, a pirymidyny (C, T) jako 0s. Lub, dwie zasady G i T są przypisane jako 1s, a pozostałe dwie A i C są 0s. Jak pokazano na rysunku 4, w celu zakodowania danych, ciąg tekstowy o długości 24 bajtów („cyfrowe przechowywanie danych DNA”) przekształcono na bity binarne, które następnie zakodowano w oligo DNA, stosując 1 bit na zasadę, przy czym purynie (A, G) przypisano jako 1s, a pirymidynie (C, T) jako 0s. Oligo DNA zostało zsyntetyzowane chemicznie, a zawartość tekstu została zapisana jako fragmenty oligo DNA w celu długoterminowego przechowywania. Dla datadecoding, fragmenty DNA były amplifikowane przez PCR, sekwencjonowane i dekodowane do bitów binarnych raz dziennie, musimy pobrać dane w celu wyjścia danych binarnych, aby być czytelne. Ostatecznie, czytanie danych z biblioteki sekwencji DNA jest do sekwencjonowania unikalnych cząsteczek DNA, konwersji informacji sekwencjonowania do oryginalnych danych cyfrowych w razie potrzeby lub wymagania (rysunek 5) .
Zalety nośnika danych DNA
Jak wspomniano powyżej, globalne dane są gwałtownie wzrosła w tempie wykładniczym. Tradycyjne media nie są w stanie w wystarczającym stopniu sprostać wymogom przechowywania dużych ilości danych. DNA może służyć jako możliwe medium do przechowywania danych cyfrowych, z jego potencjalnymi zaletami, takimi jak wysoka gęstość, wysoka wydajność replikacji, długoterminowa trwałość i długoterminowa stabilność (https:// www.scientificworldinfo.com) . DNA przy swojej teoretycznej maksymalnej pojemności może zakodować około dwóch bitów na nukleotyd. Całe centrum danych zbudowane przez IBM w 2011 roku ma około 100 petabajtów (PBs) pojemności do przechowywania danych. Jednak ze względu na dużą gęstość, DNA jako nośnik danych może przechowywać duże ilości danych przy niewielkich rozmiarach. Jeden gram DNA w swoim teoretycznym maksimum może przechowywać około 200 PB danych, czyli prawie dwa razy więcej niż całe centrum danych IBM. Innymi słowy, wszystkie informacje zapisane na całym świecie mogą być przechowywane w kilku kilogramach DNA, lub równe tylko jednemu pudełku po butach w porównaniu z wymogiem milionów dużych centrów przechowywania danych dla tradycyjnych mediów.
Zakodowany danymi nośnik DNA jest zdolny do długotrwałego przechowywania dzięki wysokiej trwałości. DNA może przetrwać tysiące lat w zimnych, suchych i ciemnych miejscach. Nawet w gorszym środowisku, okres półtrwania DNA wynosi do stu lat. DNA może zachować stabilność w niskiej lub wysokiej temperaturze, w szerokim zakresie od -800°C do 800°C. Nośniki DNA mogą również zabezpieczyć dane w większym stopniu niż tradycyjne cyfrowe nośniki danych. Chociaż nowych danych przybywa w tempie wykładniczym, większość z nich jest zapisywana w archiwach w celu długoterminowego przechowywania. Te zimne dane nie będą odzyskiwane natychmiast lub często wykorzystywane. Tak więc, przechowywanie ich na nośnikach DNA jest proste, wygodne i bezkosztowe. Kolejną zaletą jest to, że DNA jest wysoce konserwowane. Naturalne DNA może dokładnie replikować się z dużą wydajnością i zawsze zgodnie z zasadą parowania zasad (A z T, C z G) (Rysunek 3). Dlatego też nośnik DNA może w wysokim stopniu zachować wierność danych przez długi czas.
Wyzwania dla nośnika danych DNA
W oparciu o jego unikalne cechy i w porównaniu z tradycyjnymi nośnikami, DNA może być potencjalnym i obiecującym nośnikiem do przechowywania danych cyfrowych. Jednakże, przed DNA jest jeszcze długa droga do komercyjnego zastosowania. Wyzwania, z którymi musimy się zmierzyć, dotyczą różnych aspektów, w tym wysokich kosztów, niskiej przepustowości, ograniczonego dostępu do przechowywania danych, krótkich syntetycznych fragmentów oligo DNA, poziomu błędów w syntezie i sekwencjonowaniu .
Użycie DNA w przechowywaniu danych jest znacznie droższe niż inne tradycyjne nośniki, takie jak taśmy, dyski i HDD (hard disk drive) (https://www.scientificworldinfo.com). Obecnie kodowanie i dekodowanie danych kosztuje prawie 15.000 dolarów za megabajt (MB). Tymczasem, obecna technologia syntezy DNA jest ograniczona, tylko krótkie sekwencje oligo DNA mogą być syntetyzowane. Maksymalna długość każdego fragmentu oligo DNA jest ograniczona do kilkuset nukleotydów. Tak więc, aby przechować pojedynczy zarchiwizowany plik, szczególnie 1 duży plik może wymagać setek tysięcy oligo DNA. Ponadto, zapisywanie danych w oligo DNA i ich pobieranie z oligo DNA jest czasochłonne i wymaga wielu etapów, w tym konwersji danych do postaci binarnej, kodowania binarnego do oligo DNA, syntezy i przechowywania sekwencji DNA oraz pobierania unikalnych sekwencji z biblioteki do przechowywania DNA, sekwencjonowania i dekodowania, a w końcu konwersji binarnej do postaci czytelnej dla danych. Tradycyjne nośniki takie jak dyski i taśmy posiadają logiczną informację adresową, jednak oligo DNA jej nie posiada. Dlatego bardzo trudno jest zaadresować unikalną zakodowaną sekwencję DNA, którą spodziewamy się mieć. Tymczasem, losowy dostęp do przechowywania danych opartych na DNA jest ważny, jednak oligo DNA nie mają zdolności losowego dostępu. Poprzez obecne podejścia, tylko masowy dostęp jest dostępny dla przechowywania danych DNA. Cały magazyn danych oparty na DNA musi być sortowany, sekwencjonowany i dekodowany z magazynu danych DNA, nawet jeśli potrzebujemy tylko odczytać pojedynczy bajt. Dlatego wymagany jest odpowiedni primer używany do selektywnego pobierania właściwej sekwencji DNA. Zapewni to również losowy dostęp podczas sekwencjonowania DNA i pobierania danych. Sekwencjonowanie z unikalnym primerem może selektywnie odczytywać tylko wymagane oligoDNA, a nie całą bibliotekę DNA. A obecnie synteza DNA i sekwencjonowanie nie są całkowicie doskonałe. Podczas syntezy i sekwencjonowania DNA może dojść do insercji, delecji, substytucji i innych błędów, przy czym wskaźnik błędu wynosi około 1% na nukleotyd. Technologia i koszt syntezy i sekwencjonowania DNA nie są odpowiednie dla obecnego przechowywania danych.
Respective for DNA data storage medium
Due to the exponential increase of global data, lack of sufficient storage spaces and the requirement of innovative storage approaches, DNA as a potential brand new medium is becoming a hot topic in the field of big data storage. Dzięki wysokiej gęstości, wysokiej wydajności replikacji, długoterminowej trwałości i stabilności, DNA wykazuje swoje własne zalety w porównaniu z tradycyjnymi mediami do przechowywania danych. Tymczasem zastosowania cyfrowego przechowywania danych DNA były ograniczone ze względu na wysoki koszt, brak możliwości dostępu losowego, czasochłonność kodowania i dekodowania danych. Na szczęście, postęp w dziedzinie technologii DNA szybko posuwa się naprzód. Na przykład, aby zakończyć sekwencjonowanie pierwszego ludzkiego genomu, światowi naukowcy współpracowali i pracowali razem przez około 10-20 lat, a całkowity koszt wyniósł 3 miliardy dolarów w 2013 roku (strona internetowa Human Genome Project (HGP): https://www.genome.gov/human-genomeproject).
Wniosek
W dzisiejszych czasach naukowcy potrzebują zaledwie kilku tysięcy dolarów i kilku tygodni, aby zakończyć sekwencjonowanie jednego całego ludzkiego genomu. I oczekuje się, że w niedalekiej przyszłości sekwencjonowanie jednego ludzkiego genomu będzie kosztować sto dolarów lub mniej za kilka godzin. Tak więc, można oczekiwać, że koszt będzie przystępny. Dla losowego dostępu i adresowania informacji, naukowcy rozwiązali to wyzwanie poprzez zaprojektowanie unikalnych starterów, aby selektywnie adresować i pobierać wymagane informacje. W celu uniknięcia występowania błędów, metadane korekcji błędów są zakodowane we fragmentach oligo DNA. W międzyczasie zostały wynalezione i są obecnie dostępne sekwencery jednomolekularne DNA. Są one poręczne i przenośne. Mogą one jeszcze bardziej obniżyć koszty sekwencjonowania DNA i uprościć wyszukiwanie informacji o DNA. Tak więc, podążając za postępem w technologiach przechowywania danych DNA, DNA służący jako nośnik danych będzie złotą szansą w tej erze big data.
- Mayer C, McInroy GR, Murat P, Delft PV, Balasubramanian S (2016) An epigenetics-inspired DNA-based data storage system. Angew Chem Int Ed Engl 55: 11144-11148.
- Swati A, Mathuria, F, Bhavani, S, Malathy E, Mahadevan R (2017) A review on various encoding schemes used in digital DNA data storage. Int J Civil Eng Technol 8: 7-10.
- Appuswamy RLK, Barbry P, Antonini M, Madderson O, Freemont P (2019) Archive: Wykorzystanie DNA w hierarchii przechowywania DBMS. CIDR 2019, Biennal Conference on Innovative Data Systems Research, California, USA.
- De Silva PY, GU Ganegoda (2016) New trends of digital data storage in DNA. Biomed Res Int pp: 8072463-8072472.
- Panda DM, Baig KA, Swain MJ, Behera A, Dash D (2018) DNA as a digital information storage device: hope or hype? Biotech 8: 9-15.
- Chen K, Kong J, Zhu J, Ermann N, Predki P, et al. (2019) Digital data storage using DNA nanostructures and solid-state Nanopores. Nano Lett 19: 1210-1215.
- Yazdi S, Gabrys R, Milenkovic O (2017) Portable and error-free DNA-based data storage. Sci Rep 7: 5011-5013.
- Church GM, Gao Y, Kosuri S (2012) Next-generation digital information storage in DNA. Science 337: 1628-1630.
- Kuang SY, Zhu G, Wang ZL (2018) Triboelectrification-Enabled Self-Powered Data Storage. Adv Sci (Weinh) 5: 1700658.
- Block FE (1987) Analog and digital computer theory. Int J Clin Monit Comput 4: 47-51.
- O’ Driscoll A, Sleator RD (2013) Synthetic DNA: the next generation of big data storage. Bioengineered 4: 123-1235.
- Portin P (2014) Narodziny i rozwój teorii dziedziczenia DNA: sześćdziesiąt lat od odkrycia struktury DNA. J Genet 93: 293-302.
- Leu K, Obermayer B, Rajamani S, Gerland U, Chen IA (2011) The prebiotic evolutionary advantage of transferring genetic information from RNA to DNA. Nucleic Acids Res 39: 8135-8147.
- Burgers PMJ, Kunkel TA (2017) Eukaryotic DNA replication Fork. Annu Rev Biochem 86: 417-438.
- Akram F, Haq I, Ali H, Laghari AT (2018) Trends to store digital data in DNA: an overview. Mol Biol Rep 45: 1479-1490.
- Organick L, Ang SD, Chen YJ, Lopez R, Yekhanin S, et al. (2018) Random access in large-scale DNA data storage. Nat Biotechnol 36: 242-248.
- Bornholt J, Lopez R, Carmean DM, Ceze L, Seelig G, et al. (2016) A DNA-based archival storage system. ASPLOS 201 (21st ACM International Conference on Architectural Support for Programming Languages and Operating Systems, Atlanta, GA).
- Newman S, Stephenson AP, Willsey M, Nguyen BH, Takahashi CN, et al. (2019) High density DNA data storage library via dehydration with digital microfluidic retrieval. Nat Commun 10: 1706-1710.
- Yazdi SM, Yuan Y, Ma J, Zhao H, Milenkovic O (2015) A rewritable, random-access DNA-based storage system. Sci Rep 5: 14138-14140.
- Ahn T, Ban H, Park H (2018) Storing digital information in the long read DNA. Genomics Inform 16: e30-35.
- Bayley H (2017) Single-molecule DNA sequencing: Getting to the bottom of the well. Nat Nanotechnol 12: 1116-1117.
.