Health Science Journal
Palavras-chave
Armazenamento digital de dados; Ácido desoxirribonucleico (DNA); Binário; Codificação; Descodificação; Sequenciação
Introdução
Agora atual, é uma era explosiva de grandes dados. Estes grandes dados existem e cobrem quase todos os lugares, de mercearias a bancos, de offline a online, da academia à indústria, do hospital à comunidade, da organização ao governo. O grande armazenamento e gestão de dados está se tornando uma séria preocupação. Atualmente, a maioria dos dados em todo o mundo são armazenados principalmente em mídias magnéticas e ópticas, como HDD (disco rígido), DISKs, CDs, fitas, DVDs, discos rígidos portáteis e unidades USB streak. No entanto, a velocidade crescente destes dados de arquivo aumenta explosivamente a uma taxa exponencial. Essas mídias tradicionais e sua limitada capacidade de armazenamento de dados não podem atender aos requisitos do rápido aumento dos dados digitais. Entretanto, a durabilidade do armazenamento de dados dessas mídias é um grande desafio. A sua durabilidade é muito limitada. Estes meios duram apenas por um tempo muito limitado. Por exemplo, os discos podem durar vários anos e as fitas duram várias décadas. Outros suportes electrónicos podem ser mantidos em boas condições durante várias décadas. A capacidade de armazenamento de dados é outro problema para o armazenamento de grandes dados digitais. Um CD pode armazenar várias centenas de megabytes (MBs) de dados. Um disco rígido grande pode armazenar alguns terabytes (TBs) de dados. No entanto, a sua capacidade está longe da exigência dos dados de informação explosivos .
Como disse Patrizio, existem 33 Zettabytes (ZBs) de dados no mundo inteiro em 2018 (https://www.networkworld.com), equivalentes a 22 triliões de gigabytes (GBs). Portanto, uma nova tecnologia de armazenamento e um sistema inovador são necessários para atender às exigências desta era moderna. O ácido desoxirribonucleico (ADN), devido às suas vantagens únicas, deve ser o meio ideal para o armazenamento digital de dados. Armazenar os dados digitais no ADN não é uma história nova. Na verdade, ele foi descrito pelo físico soviético Mikhail Neiman em 1960 (https:// www.geneticsdigest.com). No entanto, foi a primeira vez que demonstrou que o DNA pode armazenar dados digitais em 1988 . Aqui, primeiramente introduzimos as aplicações do DNA como um novo meio de armazenamento de dados digitais e em seguida discutiremos mais detalhes neste campo do DNA servindo como meio de armazenamento de dados.
Revisão de Estudos Anteriores
O sistema numérico binário
Computadores e outros dispositivos eletrônicos digitais armazenam dados e operam com o sistema numérico binário que usa apenas dois números digitais ou 0 e 1 . Os textos são convertidos para a versão binária no sistema de computador. Por sua vez, os computadores operam, e calculam em binário, eventualmente convertem informações em textos legíveis. Um byte contém oito bits que consistem de 0 ou 1 e tem 28 (256) valores possíveis (de 0 a 255), e armazena uma única letra (Figura 1 e Tabela 1) . Como mostrado na tabela ASCII de conversão (Tabela 1). As vinte e seis letras com as letras maiúsculas e minúsculas são convertidas entre Letra, Binário e Hexadecimal. Para armazenar um arquivo ou documento grande é necessário muito mais dados de memória. Uma música normal pode precisar de dezenas de megabytes, com alguns gigabytes para armazenar um filme e vários terabytes para os livros armazenados em uma grande biblioteca. Como mostrado na Tabela 2 são os tamanhos de medida e memória para o uso do sistema binário desde a menor unidade “byte” até as unidades grandes, incluindo byte (B), kilobyte (KB), megabyte (MB), gigabyte (GB), terabyte (TB), pegabyte (PB), Exabyte (EB), zettabyte (ZB), yottabyte (YB), brontobyte (BB), Geopbyte (GPB) e assim por diante (https://www.geeksforgeeks.org&https://whatsabyte.com). As unidades como brontobyte (BB), Geopbyte (GPB) são valores enormes inimagináveis que podem nunca ser usados em nosso mundo real (Tabela 2).

p>Figure 1: A cadeia de texto “armazenamento de dados digitais de DNA” foi convertida como bits binários.
Table 1: The conversion ASCII table of the twenty-six letters with the upper and lower cases among letter, binary and hexadecimal.
| Letter | Binary | Hexadecimal | Letter | Binary | Hexadecimal |
|---|---|---|---|---|---|
| A | 1000001 | 41 | a | 1100001 | 61 |
| B | 1000010 | 42 | b | 1100010 | 62 |
| C | 1000011 | 43 | c | 1100011 | 63 |
| D | 1000100 | 44 | d | 1100100 | 64 |
| E | 1000101 | 45 | e | 1100101 | 65 |
| F | 1000110 | 46 | f | 1100110 | 66 |
| G | 1000111 | 47 | g | 1100111 | 67 |
| H | 1001000 | 48 | h | 1101000 | 68 |
| I | 1001001 | 49 | i | 1101001 | 69 |
| J | 1001010 | 4A | j | 1101010 | 6A |
| K | 1001011 | 4B | k | 1101011 | 6B |
| L | 1001100 | 4C | l | 1101100 | 6C |
| M | 1001101 | 4D | m | 1101101 | 6D |
| N | 1001110 | 4E | n | 1101110 | 6E |
| O | 1001111 | 4F | o | 1101111 | 6F |
| P | 1010000 | 50 | p | 1110000 | 70 |
| Q | 1010001 | 51 | q | 1110001 | 71 |
| R | 1010010 | 52 | r | 1110010 | 72 |
| S | 1010011 | 53 | s | 1110011 | 73 |
| T | 1010100 | 54 | t | 1110100 | 74 |
| U | 1010101 | 55 | u | 1110101 | 75 |
| V | 1010110 | 56 | v | 1110110 | 76 |
| W | 1010111 | 57 | w | 1110111 | 77 |
| X | 1011000 | 58 | x | 1111000 | 78 |
| Y | 1011001 | 59 | y | 1111001 | 79 |
| Z | 1011010 | 5A | z | 1111010 | 7A |
*Note: ACSII (American Standard Code for Information Interchange): serial digital codes to represent number, letters, numerals, and other symbols and to be used as a standard format in the computer system.
Table 2: The sizes of measurement and memory.
| Sizes | Byte Magnitude | Units | Storage* |
|---|---|---|---|
| 1 B | 100 | Byte | A character “A”, “1”, “$” |
| 10 B | 101 | ||
| 100 B | 102 | ||
| 1 KB | 103 | Kilo byte | The size for graphics of small websites ranges between 5 and 100 KB |
| 10 KB | 104 | ||
| 100 KB | 105 | ||
| 1 MB | 106 | Mega byte ( 1 MB: 1 million) |
The size for a high resolution JPEG image is about 1-5 MB |
| 10 MB | 107 | The size for a 3-minute song is about 30 MB | |
| 100 MB | 108 | ||
| 1 GB | 109 | Giga byte | The size for a standard DVD drive is about 5 GB |
| 10 GB | 1010 | (1 GB: 1 billion) | |
| 100 GB | 1011 | ||
| 1 TB | 1012 | Tera byte (1 TB: 1 trillion) |
The size for a typical internal HDD is about 2 TB |
| 10 TB | 1013 | ||
| 100 TB | 1014 | ||
| 1 PB | 1015 | Peta byte (1 PB: 1 quadrillion) |
Google store over 100 PB of all data in their drivers. |
| 10 PB | 1016 | ||
| 100 PB | 1017 | ||
| 1 EB | 1018 | Exa byte (1 EB: 1 quintillion) |
Several hundred EBs of data are transferred over global internet per year Facebook built an entire data center to store 1 EB of data in 2013 |
| 10 EB | 1019 | ||
| 100 EB | 1020 | ||
| 1 ZB | 1021 | Zetta byte (1 ZB: 1 sextillion) |
33 ZBs of global data in 2018. 160-180 ZBs of data is predicted in 2025. |
| 10 ZB | 1022 | ||
| 100 ZB | 1023 | ||
| 1 YB | 1024 | Yotta byte (1 YB: 1 septillion) |
1YB = 1 million EBs 1 YB = Size of the entire World Wide Web |
| 10 YB | 1025 | ||
| 100 YB | 1026 | ||
| 1 BB | 1027 | Bronto byte (1 BB: 1 octillion) |
1BB equals to 1 million ZBs The only thing there is to say about a Brontobyte is that it is a 1 followed by 27 zeros! |
| 10 BB | 1028 | ||
| 100 BB | 1029 | ||
| 1 GPB | 1030 | Geop byte (1 GPB: 1 nonillion) | 1, No one knows why this term was created. It is highly doubtful that anyone alive today will EVER see a Geopbyte hard drive. |
The digital data storage
Digital Data Storage (DDS) was introduced and developed in 1980s. It is a computer-based data storage technology that is based on the Digital Audio Tape (DAT) format. These digital data were stored on the silicon-based chips. Silicon is the primary material of most semiconductor and microelectronic chips. O silício puro de grau de memória raramente é encontrado na natureza. Espera-se que todo o silício de grau microchip a nível mundial se esgote num futuro próximo. Além disso, a Lei de Moore (O número de transistores acomodados nos circuitos integrados é quase o dobro a cada dois anos, ou mais chips de transistores funcionam mais rápido com mais transistores) está chegando ao fim. Assim, os chips não podem acomodar transistores adicionais e atingirão o limite de sua capacidade.
Mean enquanto isso, a maioria dos dados digitais atuais são armazenados nos tradicionais meios magnéticos, ópticos e outros como HDD (unidade de disco rígido) e CDs. Além da sua limitada capacidade de armazenamento de dados, estas mídias também podem ser mantidas por um tempo muito limitado. Eles são sensíveis ao meio ambiente ou ao estado de economia de dados. Qualquer alteração ambiental e condicional, como exposição magnética, alta umidade, alta temperatura, danos mecânicos, pode possivelmente resultar em danos a essas mídias ou na perda de dados. E o uso frequente também pode levar à sua danificação ou perda de dados. E também, para armazenar a grande quantidade de dados e para atender à exigência de aumento explosivo de dados, precisamos de uma grande quantidade de mídias como discos, CD, DVP, discos rígidos . Estes levarão a um custo elevado e serão demorados.
Simultâneamente, o aumento de dados digitais e a necessidade de armazenamento de dados estão a crescer a uma taxa exponencial. A IBM construiu um grande centro com a capacidade de armazenamento de dados de 120 PBs em 2011. O Facebook construiu outro centro maior com a capacidade de armazenamento de 1 EB (1000 PBs) de dados em 2013. Todos os usuários digitais em todo o mundo produziram mais de 44 exabytes (EBs) (44000 PBs) de dados por dia. Foram totalmente 1 zettabyte (ZB, 1000EBs ou 1 milhão de PBs) de dados produzidos globalmente em 2010 e 33 zettabytes (ZB) de dados em 2018, com 150-200 zettabytes (ZBs) sendo previstos em 2025 (citado do site Datanami: https:// www.datanami.com e do site Network world: https:// www.networkworld.com) . Para armazenar estes dados seriam necessárias centenas de milhares de enormes centros espaciais. Em 2018, o Facebook tinha um total de 15 locais de centros de dados em 2018, com mais centros novos sendo anunciados. Eles construirão quatro centros de dados adicionais no Nebraska, consistindo de seis grandes edifícios com espaço de armazenamento de dados acima de 2,6 milhões de pés quadrados. Seja o que for, o espaço de datastoring nunca poderá captar o aumento exponencial dos dados. Os meios de armazenamento atuais também não podem satisfazer a necessidade de armazenamento. Há uma necessidade urgente de desenvolver uma nova geração de tecnologia de armazenamento de dados, em vez do atual armazenamento de dados baseado em silício. Com suas características únicas e vantagens potenciais, o ácido desoxirribonucleico (DNA) como possível meio de armazenamento de dados digitais está chegando ao estágio central do armazenamento de dados.
A informação básica do ácido desoxirribonucleico (DNA)
Em 1953, o Dr. Crick e Watson revelaram que uma molécula de DNA tem fios duplos que se enrolam ao redor um do outro e formam uma estrutura helicoidal dupla. Geralmente, os materiais genéticos na maioria dos organismos naturais são filamentos duplos de DNAs helicoidais, sendo alguns filamentos simples de DNA e outros filamentos simples ou duplos de RNAs. Os componentes do DNA ou Nucleotídeos consistem em bases nitrogenadas, grupos fosfato e grupos desoxirribose. As duas letras são estruturadas como a espinha dorsal de cada molécula de DNA, com cada par de bases de cada filamento a ligar por uma ligação de hidrogênio. Os nucleotídeos de DNA consistem em quatro tipos de bases incluindo adenina (A), citosina (C), guanina (G) e timina (T) (Figura 2) , com ácido ribonucleico (RNA) tendo quatro tipos de bases incluindo adenina (A), citosina (C), guanina (G) e uracil (U) em vez de timina (T). Adenina (A) e guanina (G) são purinas, com citosina (C), tiamina (T) e uracilo (U) sendo pirimidina . In DNA molecules, the base-pairing rule is that A pairs with T, and G pairs with C (Figures 3 and 4) .
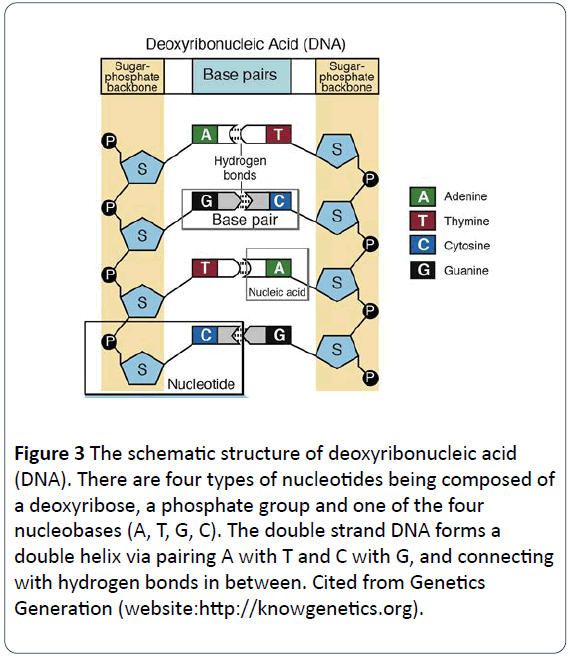
Figure 3: The schematic structure of deoxyribonucleic acid (DNA). There are four types of nucleotides being composed of a deoxyribose, a phosphate group and one of the four nucleobases (A, T, G, C). The double strand DNA forms a double helix via pairing A with T and C with G, and connecting with hydrogen bonds in between. Cited from Genetics Generation (website:https://knowgenetics.org).
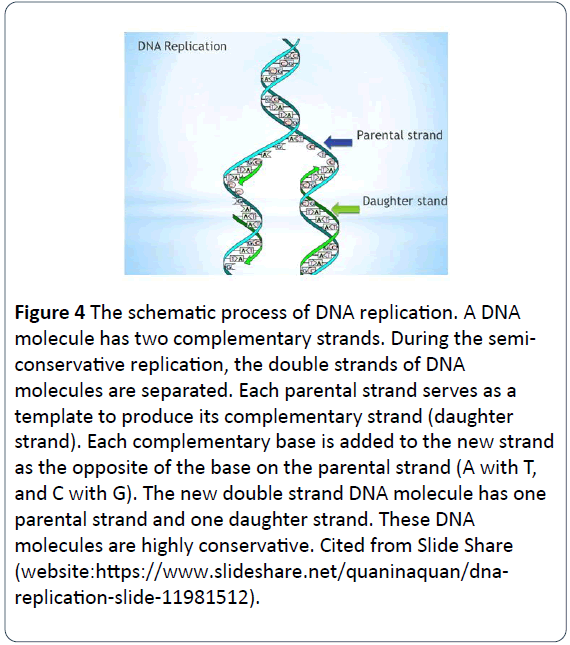
Figure 4: O processo esquemático de replicação de DNA. Uma molécula de ADN tem duas vertentes complementares. Durante a replicação semiconservadora, as fitas duplas das moléculas de DNA são separadas. Cada fita parental serve como um modelo para produzir a sua fita complementar (fita filha). Cada base complementar é adicionada à nova fita como o oposto da base sobre a fita parental (A com T, e C com G). A nova molécula de DNA de dupla fita tem uma fita parental e uma fita filha. Estas moléculas de ADN são altamente conservadoras. Citado de Slide Share (website:https://www.slideshare.net/quaninaquan/dnareplication- slide-11981512).
O processo é que uma molécula de DNA de cadeia dupla se desenrola com cada uma das duas cordas parentais separadas e atua como um modelo parental para a síntese de novas moléculas de DNA filhas. Os nucleotídeos complementares são adicionados à cadeia filha, com fosfatos e desoxirriboses para formar a espinha dorsal dos novos nucleotídeos e novas bases para emparelhar com o oposto das bases na cadeia parental através da regra de emparelhamento de bases (um emparelhamento com T, e G emparelhamento com C) e para se manter no lugar com ligações de hidrogênio . Eventualmente, cada uma das novas moléculas de DNA de cadeia dupla tem uma cadeia parental e uma cadeia filha. As moléculas de DNA replicam-se neste modelo semi-conservador, mantêm os DNAs genéticos conservadores e constantes, e passam de uma geração para outra (Figura 5) .
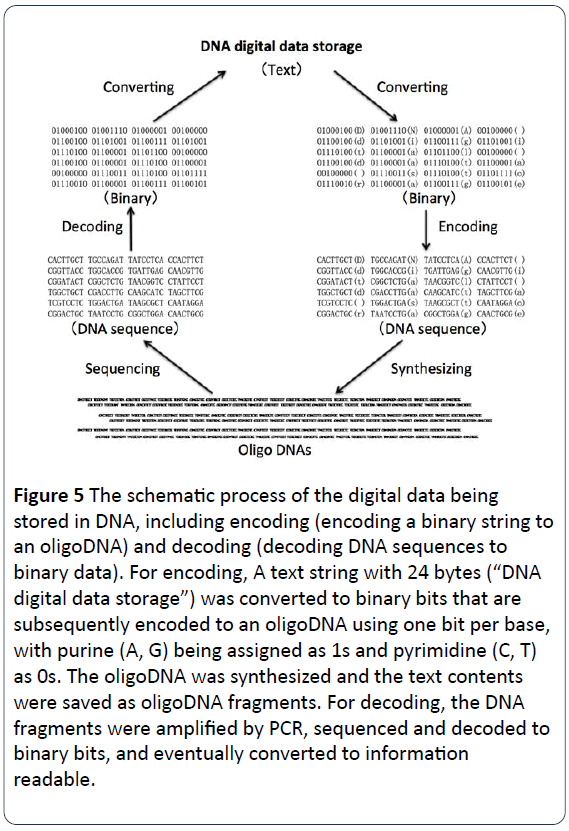
Figure 5: O processo esquemático dos dados digitais sendo armazenados no DNA, incluindo a codificação (codificando uma cadeia binária para um oligoDNA) e a decodificação (decodificando seqüências de DNA para dados binários). Para a codificação, uma cadeia de texto com 24 bytes (“DNA digital data storage”) foi convertida em bits binários que são posteriormente codificados para um oligoDNA usando um bit por base, com purine (A, G) sendo atribuído como 1s e pyrimidine (C, T) como 0s. O oligoDNA foi sintetizado e o conteúdo do texto foi salvo como fragmentos de oligoDNA. Para a descodificação, os fragmentos de ADN foram amplificados por PCR, sequenciados e descodificados em bits binários, e eventualmente convertidos em informação legível.
O processo de armazenamento de dados digitais de ADN
O processo de armazenamento de dados digitais de ADN é a codificação e descodificação de dados binários de e para as vertentes de ADN sintetizadas. Os textos, números, imagens e outros textos legíveis ou visíveis são convertidos primeiro em linguagens binárias com 0 e 1, e depois codificados em sequências nucleotídicas de ADN, com as quatro bases (A, C, G, T) em vez de 0 e 1 . Por exemplo, uma letra maiúscula “D” é “01000100” em binário, uma letra minúscula “d” é “01100100” com um branco ” ” é “00100000” (Figura 1). Nas Figuras 3 e 5, a frase “DNA digital data storage” foi convertida em versão binária para obter códigos binários com 24 bytes. Em seguida, os códigos binários (bits binários) são codificados em códigos de ADN. Cada uma das quatro bases (A, C, G e T) deve ser atribuída como 1 ou 0. Por exemplo, a purina (A, G) é atribuída como 1s, sendo a pirimidina (C, T) como 0s. Ou, as duas bases G e T são atribuídas como 1s, sendo as outras duas A e C como 0s. Como mostrado na Figura 4, para codificação de dados, uma cadeia de texto com 24 bytes (“DNA digital data storage”) foi convertida em bits binários que são posteriormente codificados em um DNA oligo usando um 1 bit por base, com purina (A, G) sendo atribuído como 1s e pirimidina (C, T) como 0s. O DNA oligo foi sintetizado quimicamente e o conteúdo do texto foi então salvo como fragmentos de DNA oligo para o armazenamento a longo prazo. Para a codificação dos dados, os fragmentos de ADN foram amplificados por PCR, sequenciados e descodificados em bits binários uma vez por dia, precisamos de recuperar os dados para que os dados binários possam ser lidos. Eventualmente, ler os dados da biblioteca de sequências de ADN é sequenciar as moléculas de ADN únicas, converter a informação sequencial em dados digitais originais conforme necessário ou requerido (Figura 5) .
As vantagens do meio de armazenamento de dados de ADN
Como mencionado acima, os dados globais são acentuadamente aumentados à taxa exponencial. A mídia tradicional não pode lidar suficientemente com a exigência de um grande armazenamento de dados . O DNA pode servir como um possível meio de armazenamento de dados digitais, com suas vantagens potenciais como alta densidade, alta eficiência de replicação, durabilidade a longo prazo e estabilidade a longo prazo (https:// www.scientificworldinfo.com) . O DNA em sua capacidade máxima teórica pode codificar cerca de dois bits por nucleotídeo . Um centro de dados inteiro construído pela IBM em 2011 tem cerca de 100 petabytes (PBs) de capacidade de armazenamento de dados. No entanto, devido à sua alta densidade, o DNA atuando como um meio de armazenamento de dados pode armazenar uma grande quantidade de dados em um tamanho pequeno. Um único grama de DNA no seu máximo teórico pode armazenar cerca de 200 PBs de dados, quase o dobro do que todo o centro de dados da IBM. Em outras palavras, todas as informações registradas em todo o mundo podem ser armazenadas em vários quilogramas de DNAs, ou igual a apenas uma caixa de sapatos em comparação com a exigência de milhões de grandes centros de armazenamento de dados para mídias tradicionais .
O meio de DNA codificado com dados é capaz de armazenar a longo prazo devido à sua alta durabilidade . O DNA pode durar milhares de anos em locais frios, secos e escuros. Mesmo em ambientes piores, a meia-vida do DNA é de até cem anos . O ADN pode reter estável a baixas ou altas temperaturas, com uma ampla gama de temperaturas entre -800°C e 800°C . O ADN também pode assegurar dados mais do que os tradicionais suportes digitais de dados . Embora novos dados estejam a aumentar a um ritmo exponencial, a maioria deles são guardados em arquivos para armazenamento a longo prazo . Estes dados frios não serão recuperados imediatamente ou utilizados com frequência. Assim, armazená-los em mídia de DNA é simples, conveniente e sem custo. Outra vantagem é que o DNA é altamente conservado. Os DNAs naturais podem se replicar com alta eficiência e sempre com a regra de emparelhamento de base (A com T, C com G) (Figura 3) . Assim, o meio de DNA pode manter a fidelidade dos dados por muito tempo.
Os desafios do meio de armazenamento de dados de DNA
Baseado nas suas características únicas e comparado com o meio tradicional, o DNA pode ser o meio potencial e promissor para o armazenamento de dados digitais . No entanto, ainda há um longo caminho a percorrer até que o ADN possa ser aplicado comercialmente. Os desafios com os quais temos de lidar existem em vários aspectos, incluindo alto custo, baixo rendimento, acesso limitado ao armazenamento de dados, fragmentos curtos de DNA sintético oligo, taxa de erro na síntese e sequenciamento .
O uso do DNA no armazenamento de dados é muito mais caro do que outras mídias tradicionais como fita, disco e HDD (disco rígido) (https://www.scientificworldinfo.com) . Atualmente, codificar e decodificar dados custa quase $15.000 por megabyte (MB). Entretanto, a tecnologia atual na síntese de DNA é limitada, com apenas seqüências curtas de DNA oligo a serem sintetizadas. O comprimento máximo de cada fragmento de DNA oligo é limitado a várias centenas de nucleotídeos . Assim, para armazenar um único arquivo, particularmente 1 arquivo grande pode precisar de centenas de milhares de DNAs de oligoeleotídeos. Além disso, é demorado para que os dados sejam gravados e recuperados dos DNAs oligo, com o envolvimento de várias etapas, incluindo a conversão de dados em binários, a codificação de DNA binário para DNA oligo, a sintetização e armazenamento de sequências de DNA e a recuperação de sequências únicas da biblioteca de armazenamento de DNA, a sequenciação e descodificação e, eventualmente, a conversão de binários em dados legíveis. As mídias tradicionais, como disco e fita adesiva, têm sua informação lógica de endereçamento, entretanto, os DNAs oligo não têm. Assim, é muito difícil abordar a sequência única de ADN codificada que esperamos ter . Entretanto, o acesso aleatório ao armazenamento de dados baseado em ADN é importante, no entanto, os Oligo DNAs não têm capacidade de acesso aleatório. Através das abordagens atuais, apenas o acesso em massa está disponível para o armazenamento de dados de DNA. Todo o armazenamento de dados baseado em ADN deve ser classificado, sequenciado e descodificado a partir do armazenamento de dados de ADN, apesar de apenas precisarmos de ler um único byte . Portanto, é necessário o primer certo usado para recuperar seletivamente a seqüência correta de DNA. Isto também fornecerá um acesso aleatório durante a sequência de ADN e a recuperação de dados. O sequenciamento com o primer único pode ler selectivamente apenas o oligoDNA necessário, em vez de toda a biblioteca de ADN . E actualmente, a síntese e sequenciação de ADN não são completamente perfeitas. Durante a síntese e seqüenciamento de DNA, a ocorrência de inserção, exclusão, substituição e outros erros pode ocorrer, com uma taxa de erro de cerca de 1% por nucleotídeo . A tecnologia e o custo da síntese e sequenciamento de DNA não são adequados para o armazenamento atual de dados .
Respectiva para o meio de armazenamento de dados de DNA
Devido ao aumento exponencial dos dados globais, à falta de espaços de armazenamento suficientes e à exigência de abordagens inovadoras de armazenamento, o DNA como um novo meio potencial está se tornando um tópico quente no campo do grande armazenamento de dados. Com a alta densidade, alta eficiência de replicação, durabilidade e estabilidade a longo prazo, o DNA exibe suas próprias vantagens em relação aos meios tradicionais de armazenamento de dados. Entretanto, as aplicações do armazenamento de dados digitais de ADN têm sido limitadas devido ao alto custo, à falta de capacidade de acesso aleatório, ao tempo gasto na codificação e descodificação de dados. Felizmente, o progresso no campo da tecnologia do ADN está a avançar rapidamente. Por exemplo, para completar a sequência do primeiro genoma humano, os cientistas globais colaboraram e trabalharam juntos durante cerca de 10-20 anos, com o custo total de 3 bilhões de dólares em 2013 (site do Projeto Genoma Humano (HGP)): https://www.genome.gov/human-genomeproject).
Conclusion
Agora, os cientistas só precisam de vários milhares de dólares e algumas semanas para terminar a sequência de um genoma humano inteiro. E espera-se que o sequenciamento de um genoma humano apenas custe cem dólares ou menos por várias horas num futuro próximo. Assim, pode-se esperar que o custo seja acessível. Para o acesso aleatório e o tratamento da informação, os cientistas resolveram este desafio através da concepção de cartilhas únicas para tratar e recuperar a informação necessária de forma selectiva. A fim de evitar a ocorrência de erros, os metadados de correção de erros são codificados em fragmentos de DNA oligo. Entretanto, os sequenciadores de ADN de moléculas únicas foram inventados e estão actualmente disponíveis. Eles são práticos e portáteis. Eles podem reduzir ainda mais o custo do sequenciamento de ADN e simplificar a recuperação da informação de ADN. Assim, seguindo os avanços nas tecnologias de armazenamento de dados de DNA, o DNA servindo como um meio de armazenamento de dados será uma oportunidade de ouro nesta era de grandes dados.
- Mayer C, McInroy GR, Murat P, Delft PV, Balasubramanian S (2016) Um sistema de armazenamento de dados baseado em DNA inspirado em epigenética. Angew Chem Int Ed Engl 55: 11144-11148.
- Swati A, Mathuria, F, Bhavani, S, Malathy E, Mahadevan R (2017) Uma revisão sobre vários esquemas de codificação usados no armazenamento de dados de ADN digital. Int J Civil Eng Technol 8: 7-10.
- Appuswamy RLK, Barbry P, Antonini M, Madderson O, Freemont P (2019) Arquivo: Usando o DNA na hierarquia de armazenamento do SGBD. CIDR 2019, Biennal Conference on Innovative Data Systems Research, California, USA.
- De Silva PY, GU Ganegoda (2016) Novas tendências de armazenamento de dados digitais no DNA. Biomed Res Int pp: 8072463-8072472.
- Chen K, Kong J, Zhu J, Ermann N, Predki P, et al. (2019) Armazenamento digital de dados usando nanoestruturas de DNA e nanoporos de estado sólido. Nano Lett 19: 1210-1215.
- Yazdi S, Gabrys R, Milenkovic O (2017) Armazenamento de dados baseado em DNA portátil e livre de erros. Rep. Sci 7: 5011-5013.
- Church GM, Gao Y, Kosuri S (2012) Armazenamento de informação digital de próxima geração em ADN. Science 337: 1628-1630.
- Kuang SY, Zhu G, Wang ZL (2018) Armazenamento de dados auto-electrificados com tribo-electrificação. Adv Sci (Weinh) 5: 1700658.
- Block FE (1987) Teoria de computadores analógicos e digitais. Int J Clin Monit Comput 4: 47-51.
- O’ Driscoll A, Sleator RD (2013) Synthetic DNA: a próxima geração de grande armazenamento de dados. Bioengenharia 4: 123-1235.
- Portin P (2014) O nascimento e desenvolvimento da teoria do DNA de herança: sessenta anos desde a descoberta da estrutura do DNA. J Genet 93: 293-302.
- Leu K, Obermayer B, Rajamani S, Gerland U, Chen IA (2011) A vantagem evolutiva prebiótica da transferência de informação genética do RNA para o DNA. Ácidos Nucleicos Res 39: 8135-8147.
- Hambúrgueres PMJ, Kunkel TA (2017) Garfo de replicação de DNA eucariótico. Annu Rev Biochem 86: 417-438.
- Akram F, Haq I, Ali H, Laghari AT (2018) Tendências para armazenar dados digitais no DNA: uma visão geral. Mol Biol Rep 45: 1479-1490.
- Organick L, Ang SD, Chen YJ, Lopez R, Yekhanin S, et al. (2018) Acesso aleatório no armazenamento de dados de DNA em larga escala. Nat Biotechnol 36: 242-248.
- Bornholt J, Lopez R, Carmean DM, Ceze L, Seelig G, et al. (2016) Um sistema de armazenamento de arquivos baseado em DNA. ASPLOS 201 (21ª ACM International Conference on Architectural Support for Programming Languages and Operating Systems, Atlanta, GA).
- Newman S, Stephenson AP, Willsey M, Nguyen BH, Takahashi CN, et al. (2019) Biblioteca de armazenamento de dados de DNA de alta densidade via desidratação com recuperação digital de microfluidos. Nat Commun 10: 1706-1710.
- Ahn T, Ban H, Park H (2018) Armazenamento de informação digital no ADN de leitura longa. Genomics Inform 16: e30-35.
- Bayley H (2017) Sequenciação de ADN monomolécula: Chegando ao fundo do poço. Nat Nanotechnol 12: 1116-1117.
li> Panda DM, Baig KA, Swain MJ, Behera A, Dash D (2018) DNA como dispositivo de armazenamento de informação digital: esperança ou hype? Biotech 8: 9-15.
li> Yazdi SM, Yuan Y, Ma J, Zhao H, Milenkovic O (2015) Um sistema de armazenamento de ADN regravável, de acesso aleatório. Sci Rep 5: 14138-14140.